无监督学习
聚类算法
与分类算法区别:分类算法仍是监督学习的范畴,样本数据除了特征,还带有标签;而聚类算法是一种无监督学习算法,样本数据只有特征没有标签
查看数据集中所有数据,在数据集中找到彼此相似数据组,就称为聚类
K-Means 算法
算法描述
K-Means存储用于定义聚类的 k 个质心。如果一个点离哪个质心最接近,则该点被视为位于哪个聚类中
K-Means的目标是**最小化簇内平方误差和 WCSS**:每个点到其所属簇质心的距离的平方和
WCSS=∑i=1K∑x∈c(i)∣∣x−μi∣∣2K:簇的数量c(i):第i个簇(类别)的点集μi:第i个簇(类别)的质心 WCSS=\sum_{i=1}^{K}\sum_{x \in c^{(i)}}||x-\mu_i||^2\\ K:簇的数量\\ c^{(i)}:第i个簇(类别)的点集\\ \mu_i:第i个簇(类别)的质心\\ WCSS=i=1∑Kx∈c(i)∑∣∣x−μi∣∣2K:簇的数量c(i):第i个簇(类别)的点集μi:第i个簇(类别)的质心
K-Means接受参数k,将事先输入的n个对象划分为k个聚类,以便使所获得的聚类满足:
- 同一聚类中的对象相似度较高
- 不同聚类中的对象相似度较小
K-Means通过交替进行下面两步来找到最佳质心:
- 根据当前质心将数据点划分到某聚类中
- 根据当前数据点计算聚类的质心
算法思想
以空间中k个点为中心进行聚类,对最靠近他们的对象归类。通过迭代的方法,逐次更新各聚类中心的值,直至得到最好的聚类结果
算法步骤
初始化 k 个聚类的质心μ
μ1,μ2,…,μk∈Rn \mu_1,\mu_2,\dots,\mu_k \in R^n μ1,μ2,…,μk∈Rn
重复以下两步,迭代直到收敛
对于每个数据点 xi,找到距离最近的质心对应的类别
c(i)=argminj∣∣x(i)−μj∣∣2argminarg(exp)是找到使exp最小的arg即c(i)是使∣x(i)−μj∣2最小的j c^{(i)}=argmin_j||x^{(i)}-\mu_j||^2 \\ argmin_{arg}(exp)是找到使exp最小的arg\\ 即c^{(i)}是使|x^{(i)}-\mu_j|^2最小的j c(i)=argminj∣∣x(i)−μj∣∣2argminarg(exp)是找到使exp最小的arg即c(i)是使∣x(i)−μj∣2最小的j
对每个类别 j,根据这个类别中数据的平均值计算得到新的质心
μj:=∑i=1m1{c(i)=j}x(i)∑i=1m1{c(i)=j} \mu_j := \frac{\sum_{i=1}^{m}1\{{c^{(i)}=j\}}x^{(i)}}{\sum_{i=1}^{m}1\{{c^{(i)}=j\}}} μj:=∑i=1m1{c(i)=j}∑i=1m1{c(i)=j}x(i)
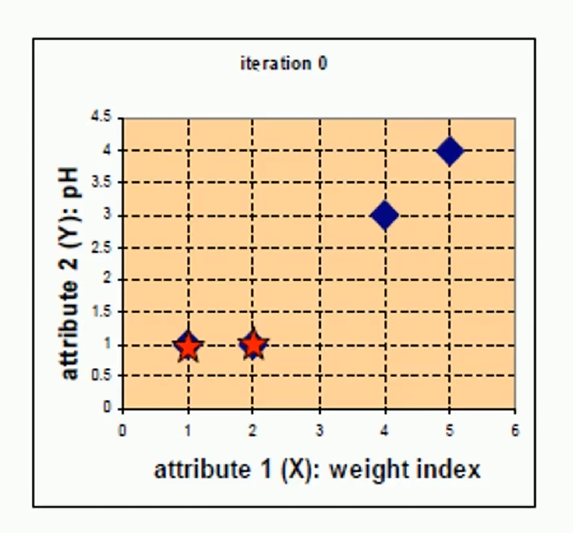
比如上面这张图
点集为[(1,1),(2,1),(4,3),(5,4)] = [A,B,C,D]
选取质心为(1,1),(2,1),那么所有点到质心的距离为
[0. 1. 3.60555128 5. ] -- group-1
[1. 0. 2.82842712 4.24264069] -- group-2
使用one-hot表示这一轮后的点的分类
[1 0 0 0]
[0 1 1 1]
A B C D
为什么这个矩阵一开始看起来这么诡异?
因为使用one-hot编码表示属于哪个质心对应的类别
更新质心
(1,1)对应的类别c1中,还是只有质心一个数据对象,不用更新
(2,1)对应的类别c2中,新增了2个数据对象,更新
μ2=(2+4+53,1+3+43)=(113,83) \mu_2 = (\frac{2+4+5}{3},\frac{1+3+4}{3})=(\frac{11}{3},\frac{8}{3}) μ2=(32+4+5,31+3+4)=(311,38)
迭代上述过程,直到聚类不发生变化
代码实现
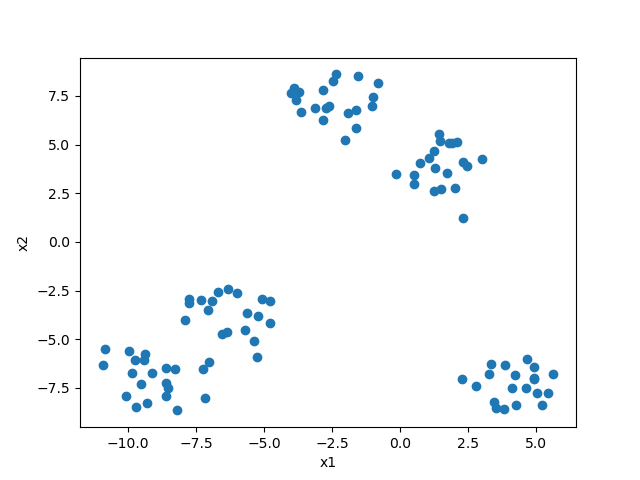
导入数据,使用sklearn.datasets.make_blobs()生成聚类数据
from sklearn.datasets import make_blobs
# 100个样本,5个聚类中心
X, y = make_blobs(n_samples=100, centers=5)
点图
初始化质心
# 初始化质心
def init_centroid(X, k):
sample_size, f_size = X.shape
# k个质心的集合
centroids = np.zeros((k, f_size))
for i in range(k):
# 随机索引
index = int(np.random.uniform(low=0, high=sample_size))
# 抽到的点加入质心集合
centroids[i, :] = X[index, :]
return centroids
K-Means
def kmeans(X, k):
sample_size = X.shape[0]
# 初始化质心
centroids = init_centroid(X, k)
# cluster_data[:,0]表示数据点所属的簇,cluster_data[:,1]表示数据点到簇质心的距离
cluster_data = np.zeros((sample_size, 2))
# 当簇是否更新的标志
flag = True
while flag:
flag = False
for i in range(sample_size):
# 最小距离
min_dist = np.inf
# 最近质心的索引
min_index = -1
for j in range(k):
diff = X[i, :] - centroids[j, :]
# print(f"diff.shape={diff.shape}")
dist = np.sqrt(np.sum(diff ** 2))
# print(f"dist={dist}")
# 更新与附近质心的最近距离
if dist < min_dist:
min_dist = dist
cluster_data[i][1] = dist
min_index = j
# 聚类发生变化
if cluster_data[i][0] != min_index:
flag = True
cluster_data[i][0] = min_index
for j in range(k):
# 类别为j的索引
index = np.nonzero(cluster_data[:, 0] == j)
# 所有类别为j的数据对象
data = X[index]
# 更新质心
centroids[j, :] = np.mean(data, axis=0)
return centroids, cluster_data
绘制聚类图
def show(X, centroids, cluster_data, k):
sample_size = X.shape[0]
# 红色圆点,蓝色圆点,绿色圆点,黄色圆点
mark = ['or', 'ob', 'og', 'oy']
# 红色星星,蓝色星星,绿色星星,黄色星星
m = ['*r', '*b', '*g', '*y']
for i in range(sample_size):
# 获取所属的簇
index = int(cluster_data[i, 0])
plt.plot(X[i, 0], X[i, 1], mark[index])
for i in range(k):
plt.plot(centroids[i, 0], centroids[i, 1], m[i], markersize=20)
plt.show()
全部代码
import numpy as np
from matplotlib import pyplot as plt
from sklearn.datasets import make_blobs
def plot_point(X):
plt.scatter(X[:, 0], X[:, 1])
plt.xlabel("x1")
plt.ylabel("x2")
plt.show()
# 初始化质心
def init_centroid(X, k):
sample_size, f_size = X.shape
# k个质心的集合
centroids = np.zeros((k, f_size))
for i in range(k):
# 随机索引
index = int(np.random.uniform(low=0, high=sample_size))
# 抽到的点加入质心集合
centroids[i, :] = X[index, :]
return centroids
def kmeans(X, k):
sample_size = X.shape[0]
# 初始化质心
centroids = init_centroid(X, k)
# cluster_data[:,0]表示数据点所属的簇,cluster_data[:,1]表示数据点到簇质心的距离
cluster_data = np.zeros((sample_size, 2))
# 当簇是否更新的标志
flag = True
while flag:
flag = False
for i in range(sample_size):
# 最小距离
min_dist = np.inf
# 最近质心的索引
min_index = -1
for j in range(k):
diff = X[i, :] - centroids[j, :]
# print(f"diff.shape={diff.shape}")
dist = np.sqrt(np.sum(diff ** 2))
# print(f"dist={dist}")
# 更新与附近质心的最近距离
if dist < min_dist:
min_dist = dist
cluster_data[i][1] = dist
min_index = j
# 聚类发生变化
if cluster_data[i][0] != min_index:
flag = True
cluster_data[i][0] = min_index
for j in range(k):
# 类别为j的索引,np.nonzero()会返回布尔数组中为True的元素的索引
index = np.nonzero(cluster_data[:, 0] == j)
# 所有类别为j的数据对象
data = X[index]
# 更新质心
centroids[j, :] = np.mean(data, axis=0)
return centroids, cluster_data
def show(X, centroids, cluster_data, k):
sample_size = X.shape[0]
# 红色圆点,蓝色圆点,绿色圆点,黄色圆点
mark = ['or', 'ob', 'og', 'oy']
# 红色星星,蓝色星星,绿色星星,黄色星星
m = ['*r', '*b', '*g', '*y']
for i in range(sample_size):
# 获取所属的簇
index = int(cluster_data[i, 0])
plt.plot(X[i, 0], X[i, 1], mark[index])
for i in range(k):
plt.plot(centroids[i, 0], centroids[i, 1], m[i], markersize=20)
plt.show()
# 100个样本,4个聚类中心
X, y = make_blobs(n_samples=100, centers=4, random_state=42)
k = 4
centroids, cluster_data = kmeans(X, k)
# 检验空簇
if np.isnan(centroids).any():
print("Error")
else:
print("Success")
show(X, centroids, cluster_data, k)
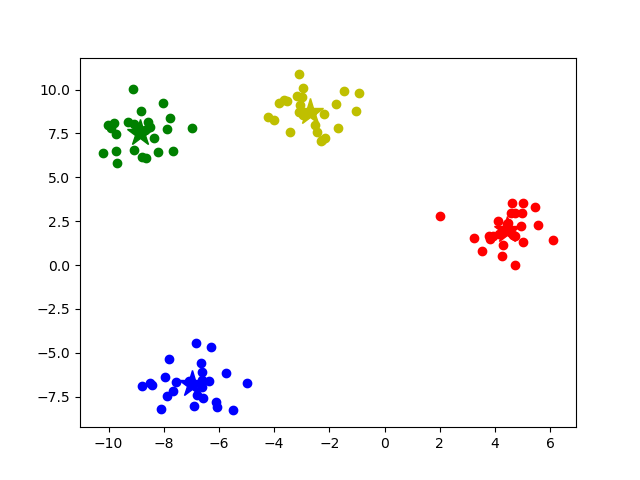
得到的聚类图
存在的问题
- 更新质心时应该是更新当前类别为j的数据对象,而不是更新所有的;传错参数最后会导致所有数据都是一个聚类
- 有时会出现簇消失的情况,最终分类的簇的个数小于我们预设的k,如图所示
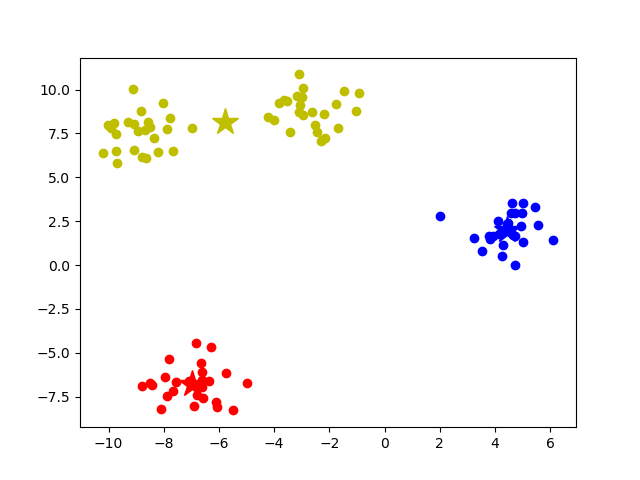
此时的质心列表
[[-6.9749469 -6.76471966]
[ 4.44441765 2.02122148]
[ nan nan]
[-5.77644235 8.14575097]]
为什么会这样呢?
因为j类簇是个空簇,所以获取j类别里元素索引列表的时候,获取的列表是空,取均值时会变成nan
出现空簇是因为:我们的质心是随机选取的,有质心重合的可能性,造成有一个质心对应的的簇变成空簇
解决:
抽取质心的时候就不让它重复
# 初始化质心:改用np.random.choice进行不充分抽样
def init_centroid(X, k):
sample_size, f_size = X.shape
# 从0-sample_size-1随机抽取k个不重复的数作为索引
index = np.random.choice(sample_size, size=k, replace=False)
centroids = X[index]
return centroids
使用sklearn的api
基本使用
from sklearn.cluster import KMeans
数据
# 100个样本,5个聚类中心
X, y = make_blobs(n_samples=100, centers=4, random_state=42)
k = 4
模型
kmeans = KMeans(
n_clusters=4, # 分为k类
init="random", # 初始随机选取质心
n_init=5, # 初始质心数
max_iter=100 # 最大迭代次数
)
拟合数据
kmeans.fit(X)
获取每个样本的聚类标签
cluster_label = kmeans.predict(X)
# 二者等价
cluster_label = kmeans.labels_
获取质心
kmeans.cluster_centers_
绘图
def fill(model, X, labels):
X_min, X_max = X[:, 0].min(), X[:, 0].max()
y_min, y_max = X[:, 1].min(), X[:, 1].max()
# 创建网格
xx, yy = np.meshgrid(
np.arange(X_min, X_max, 0.02),
np.arange(y_min, y_max, 0.02),
)
# np.c_[]表示按列拼接,比如[1,2,3]和[4,5,6]拼接后变成[[1,4],[2,5],[3,6]]
# .ravel()和.flatten()都是将多维数组展平,但是.flatten返回的是副本,.ravel()返回视图
z = model.predict(np.c_[xx.ravel(), yy.ravel()])
# 模型预测结果是1D,升维
z = z.reshape(xx.shape)
# 绘制填充等高线
cs = plt.contourf(xx, yy, z)
data_size = X.shape[0]
# 红色圆点,蓝色圆点,绿色圆点,黄色圆点
mark = ['or', 'ob', 'og', 'oy']
for i in range(data_size):
plt.plot(X[i, 0], X[i, 1], mark[labels[i]], alpha=0.3)
for i in range(k):
plt.plot(centers[i, 0], centers[i, 1], '*w', markersize=20)
plt.show()
最终聚类结果图
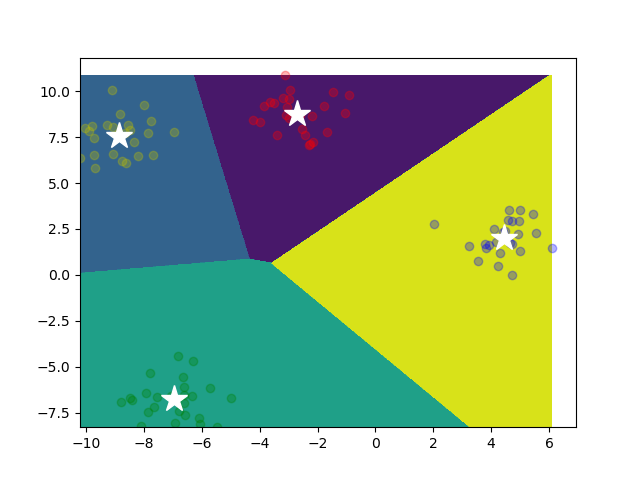
Mini Batch K-Means
Mini Batch K-Means是K-Means的一个变种;采用小批量的数据子集迭代更新聚类中心。小批量指的是每次训练算法时所随机抽取的数据子集
优点
- 只是用数据的一个小批量,计算量减少了,收敛速度加快
- 聚类结果与标准
K-Means相近,但是速度更快
迭代步骤
- 从数据集中随机抽取一些数据形成小批量,把他们分配给最近的质心
- 更新质心,与K-Means相比,
Mini Batch K-Means是在更小的样本集上更新
sklearn-api
from sklearn.cluster import MiniBatchKMeans
mini_batch_kmeans = MiniBatchKMeans(n_clusters=4)
KMeans缺点
对初始质心的选择较敏感
k值选取是由用户决定,k不一定最优
k太大
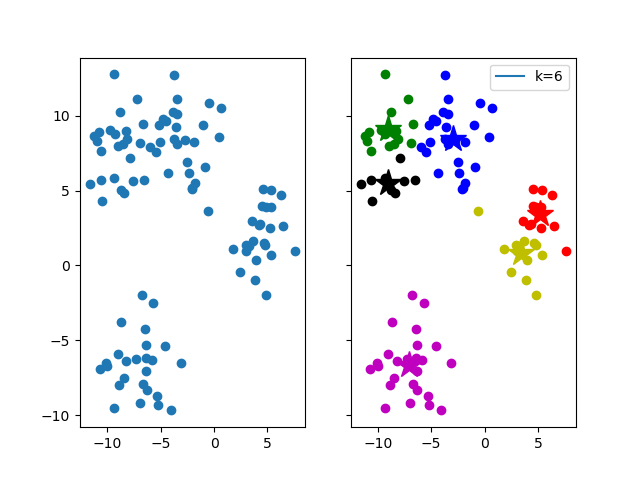
有不必要的划分
k太小
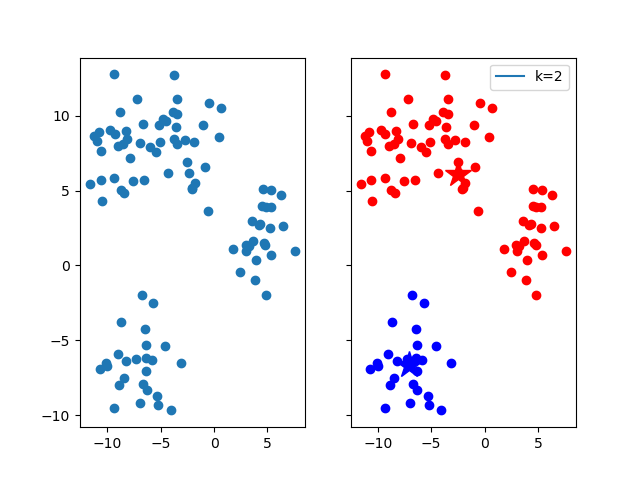
没有完全区分开聚类
不适合非球型簇,K-means 假设每个簇是球形且大小相近
用月牙形数据进行验证
from sklearn.datasets import make_moons X, y = make_moons(n_samples=100, noise=0.05, random_state=42)原数据点与聚类结果,我们预想的应该是:每个月牙形是一个聚类
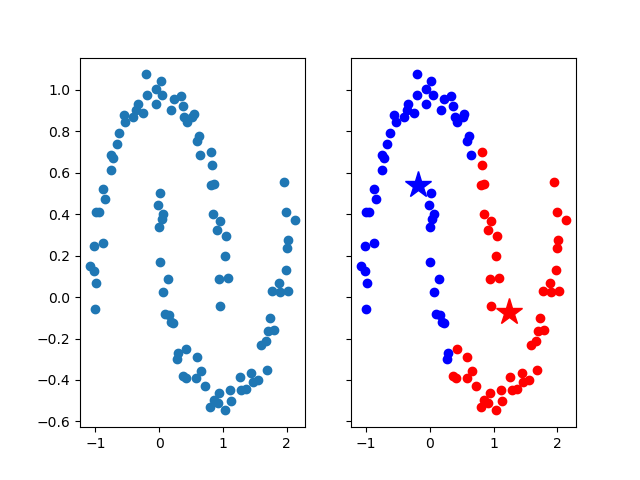
但是实际上并没有,
K-Means的搜索逻辑就是按距离搜索,所以聚类的形状会趋近于球型容易陷入局部最优
如图所示,确实是收敛了,但是只是收敛到了局部最小值
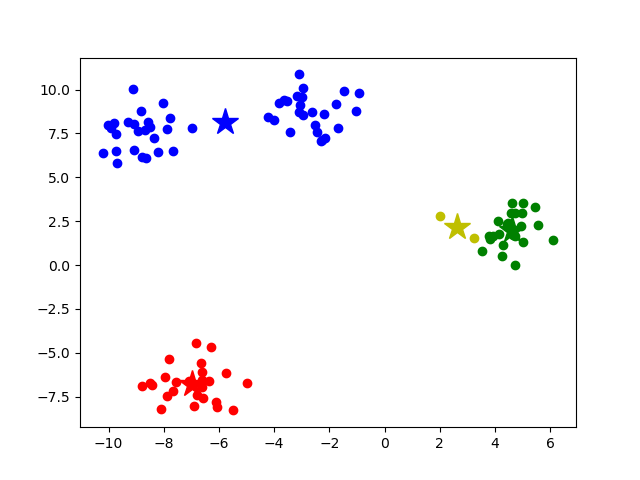
算法要求样本存在均值(为了更新质心),限制了数据的种类
对噪声敏感
使用cluster_std指定高斯分布簇的松散程度,相当于引入噪音
X, y = make_blobs( n_samples=100, centers=4, random_state=42, cluster_std=10 # 控制松散程度,越大越松散,相当于引入噪音 )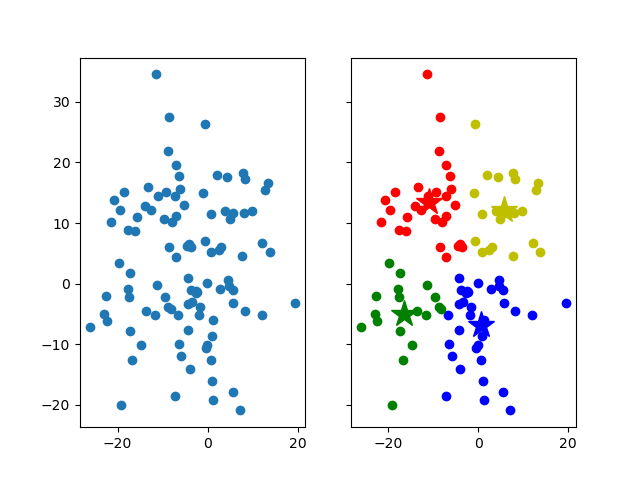
K-Means优化
对初始质心的选择
使用多次的随机初始化,计算每一次建模得到的代价函数的值,选取代价函数最小的结果作为聚类结果
代价函数/簇内平方误差和WCSS
WCSS=∑i=1K∑x∈c(i)∣∣x−μi∣∣2K:簇的数量c(i):第i个簇(类别)的点集μi:第i个簇(类别)的质心∣∣∣∣表示取模操作,方便将算法向量化 WCSS=\sum_{i=1}^{K}\sum_{x \in c^{(i)}}||x-\mu_i||^2\\ K:簇的数量\\ c^{(i)}:第i个簇(类别)的点集\\ \mu_i:第i个簇(类别)的质心\\ || ||表示取模操作,方便将算法向量化 WCSS=i=1∑Kx∈c(i)∑∣∣x−μi∣∣2K:簇的数量c(i):第i个簇(类别)的点集μi:第i个簇(类别)的质心∣∣∣∣表示取模操作,方便将算法向量化
代码实现
循环:
K-Mean利用最开始的手动实现
def cost(cluster_data, X, centers):
k = centers.shape[0]
loss = 0
for j in range(k):
index = np.nonzero(cluster_data[:, 0] == j)
cluster = X[index]
diff = cluster - centers[j]
loss += (np.sum(diff ** 2))
return loss
使用向量化进行优化
模型使用sklearn的api
def WCSS(X, labels, centers):
# 获取每个点对应的质心
# 利用numpy的整数索引,相当于assigned_centers[i]=centers[labels[i]],shape=(labels.shape[0],centers.shape[1])
assigned_centers = centers[labels]
diff = X - assigned_centers
return np.sum(diff ** 2)
模型
# 100个样本,5个聚类中心
X, y = make_blobs(
n_samples=100,
centers=4,
random_state=42,
cluster_std=2 # 控制松散程度,越大越松散,相当于引入噪音
)
k = 4
kmeans = KMeans(n_clusters=k)
kmeans.fit(X)
labels = kmeans.labels_
centers = kmeans.cluster_centers_
选出最优质心
改进后的KMeans
def kmeans(X, k, iters=100):
'''
:param X: 数据
:param k: 最终分出的聚类数
:param iters: 迭代多少次找到最优质心
:return:
'''
sample_size = X.shape[0]
best_centers = np.zeros((k, 2))
best_cluster_data = np.zeros((k, 2))
best_wcss = np.inf
for _ in range(iters):
# 当簇是否更新的标志
flag = True
# 初始化质心
centroids = init_centroid(X, k)
# cluster_data[:,0]表示数据点所属的簇,cluster_data[:,1]表示数据点到簇质心的距离
cluster_data = np.zeros((sample_size, 2))
while flag:
flag = False
for i in range(sample_size):
# 最小距离
min_dist = np.inf
# 最近质心的索引
min_index = -1
for j in range(k):
diff = X[i, :] - centroids[j, :]
# print(f"diff.shape={diff.shape}")
dist = np.sqrt(np.sum(diff ** 2))
# print(f"dist={dist}")
# 更新与附近质心的最近距离
if dist < min_dist:
min_dist = dist
cluster_data[i][1] = dist
min_index = j
# 聚类发生变化
if cluster_data[i][0] != min_index:
flag = True
cluster_data[i][0] = min_index
for j in range(k):
# 类别为j的索引
index = np.nonzero(cluster_data[:, 0] == j)
# 所有类别为j的数据对象
if len(index) > 0:
data = X[index]
# 更新质心
centroids[j, :] = np.mean(data, axis=0)
else:
print("空簇")
wcss = cost(cluster_data, X, centroids)
if wcss < best_wcss:
best_wcss = wcss
best_centers = centroids
best_cluster_data = cluster_data
return best_centers, best_cluster_data
def show(X, centroids, cluster_data, ax, k):
sample_size = X.shape[0]
# 圆点(circle)
mark = ['or', 'ob', 'og', 'oy', 'ok', 'om', 'oc'] # 红、蓝、绿、黄、黑、品红、青
# 星星(star)
m = ['*r', '*b', '*g', '*y', '*k', '*m', '*c']
for i in range(sample_size):
# 获取所属的簇
index = int(cluster_data[i, 0])
ax.plot(X[i, 0], X[i, 1], mark[index])
for i in range(k):
ax.plot(centroids[i, 0], centroids[i, 1], m[i], markersize=20)
ax.plot([], [], label=f"k={k}")
plt.legend()
plt.show()
def cost(cluster_data, X, centers):
k = centers.shape[0]
loss = 0
for j in range(k):
index = np.nonzero(cluster_data[:, 0] == j)
cluster = X[index]
diff = cluster - centers[j]
loss += (np.sum(diff ** 2))
return loss
最终聚类图
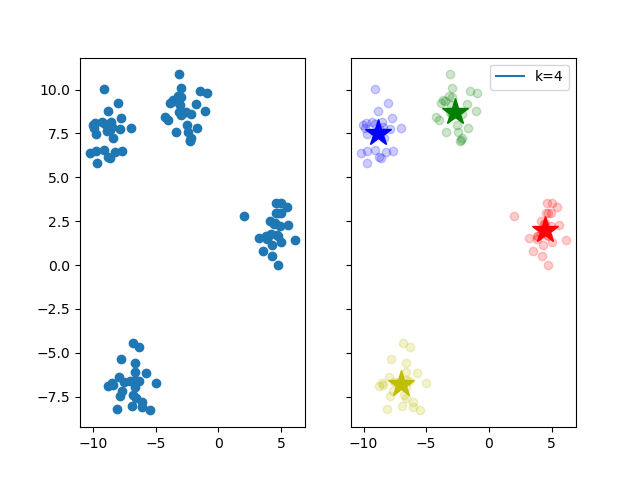
K-Means++
核心思想
让每个新的聚类中心尽可能远离已选的聚类中心
算法流程
- 随机选取一个聚类中心μ1,从数据集中随机选取一个点
- 对第i个聚类中心(i=2,…,k)
计算每个样本x到当前已选的所有聚类中心的最短距离D(x)
D(x)=minμ1,…,μi−1∣∣x−μj∣∣j=1,…,i−1 D(x)=\min_{\mu_1,\dots,\mu_{i-1}}||x-\mu_j||\\ j=1,\dots,i-1 D(x)=μ1,…,μi−1min∣∣x−μj∣∣j=1,…,i−1按照概率P(x)选择下一个聚类中心
P(x)=D(x)2∑xD(x)2 P(x) = \frac{D(x)^2}{\sum_xD(x)^2} P(x)=∑xD(x)2D(x)2
并不是选取概率最大的,而是按照概率分布选择(?)- 得到每个点的概率之后,我们需要计算累积概率,比如概率分布[0.1,0.2,0.7],累积概率分布为[0.1,0.3,1.0]
- 生成一个[0,1]之间的随机数r
- 找到第一个累积概率大于等于r的点作为下一个质心
3.一旦所有 k 个初始聚类中心选定,后续步骤与标准 K-Means 相同
sklearn中使用K-Means++
init属性默认使用k-means++;也可以构建K-Means模型时显式指定init属性为k-means++
kmeans = KMeans(
n_clusters=k, # 分为k类
init="k-means++", # 使用k-means++选取质心
n_init=5, # 初始质心数
max_iter=100 # 最大迭代次数
)
手动实现
def get_min_dists(X, centers):
'''
:param X: 数据
:param centers: 当前的所有质心
:return: 所有样本点与其最近的质心的距离
'''
sample_size = X.shape[0]
min_dists = np.zeros(sample_size)
for i in range(sample_size):
min_dist = np.inf
for center in centers:
dist = np.sum((X[i] - center) ** 2)
if dist < min_dist:
min_dist = dist
min_dists[i] = min_dist
return min_dists
def kmeans_pp(X, k):
sample_size, _ = X.shape
centers = []
# 随机选择第一个中心点
first_idx = np.random.choice(sample_size, size=1)[0]
centers.append(X[first_idx])
for _ in range(k - 1):
dists = get_min_dists(X, centers)
# 构建概率分布
probs = dists / dists.sum()
# 计算累计和
cum_probs = np.cumsum(probs)
# 使用转盘法
# 生成一个 [0,1] 之间的随机数
r = np.random.rand()
# 找到第一个累积概率大于等于 r 的样本点
for i in range(sample_size):
if cum_probs[i] >= r:
centers.append(X[i])
break
return np.array(centers)
K值选取
肘部法则
绘制不同k值下WCSS的图像,寻找"肘部"点作为最佳K值
核心思想
随着k值的增大,聚类的误差会不断减小,但当k增大到某个值之后,误差下降的幅度会显著变小,形成一个肘部形状的拐点
绘图代码
K-Means使用前面对选取初始质心优化后的代码
def manba(Ks):
X, y = make_blobs(n_samples=300, centers=4, random_state=42)
wcss = []
for k in Ks:
centroids, cluster_data = kmeans(X, k, iters=10)
current_wcss = cost(cluster_data, X, centroids)
wcss.append(current_wcss)
plt.figure(figsize=(8, 5))
plt.plot(Ks, wcss, 'bo-', linewidth=2, markersize=8)
plt.title('Elbow Method for Optimal k')
plt.xlabel('Number of clusters (k)')
plt.ylabel('Within-Cluster Sum of Squares (WCSS)')
plt.grid(True)
# 用k的取值作为横轴刻度
plt.xticks(Ks)
plt.show()
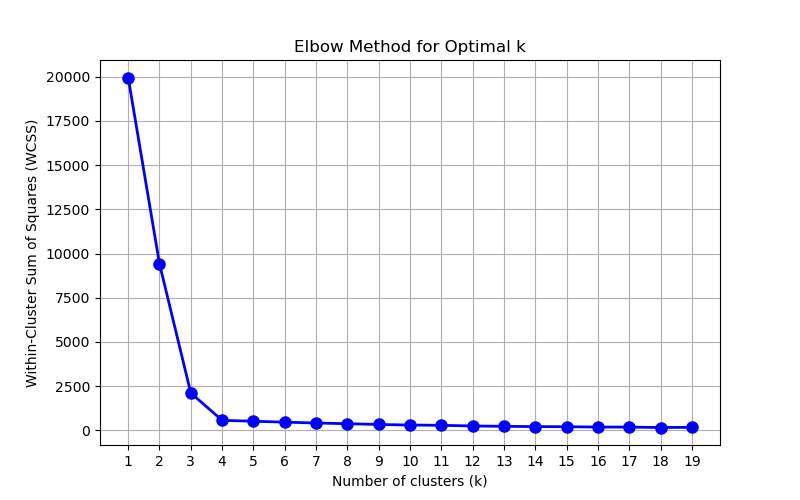
由图中我们可以看到k=4是拐点,而我们创建的数据集设置簇为4,所以最终聚类聚成4个类最合适
缺点:主观性较强,尤其是如果肘部不明显
轮廓系数
用于衡量聚类结果中每个样本的“聚类合理性”,即该样本是否被正确分配到正确的簇中
对于每个样本点i,其轮廓系数s(i)的计算公式如下:
s(i)=b(i)−a(i)max{a(i),b(i)}a(i):样本点i到同簇其他点的平均距离b(i):样本点i到最近其他簇中所有点的平均距离 s(i)= \frac{b(i)-a(i)}{max\{a(i),b(i)\}} \\ a(i):样本点i到同簇其他点的平均距离\\ b(i):样本点i到最近其他簇中所有点的平均距离\\ s(i)=max{a(i),b(i)}b(i)−a(i)a(i):样本点i到同簇其他点的平均距离b(i):样本点i到最近其他簇中所有点的平均距离
轮廓系数的取值范围位于[-1,1]
- -1:样本点可能错误地被分配到了其他簇
- 0:样本点位于两个簇之间
- 1:样本点被聚类得很好
使用sklearn实现
# 计算整体轮廓系数和单个样本轮廓系数
from sklearn.metrics import silhouette_score,silhouette_samples
寻找最优的k:轮廓系数最大对应的k
def get_best_K(Ks):
X, y = make_blobs(
n_samples=100,
centers=4,
random_state=42,
)
plt.scatter(X[:, 0], X[:, 1], c='b')
scores = []
for k in Ks:
kmeans = KMeans(n_clusters=k)
kmeans.fit(X)
labels = kmeans.labels_
score = silhouette_score(X, labels)
print(score)
scores.append(score)
plt.figure(figsize=(8, 5))
plt.plot(Ks, scores, 'bo-', linewidth=2, markersize=8)
plt.xlabel('Number of clusters (k)')
plt.ylabel('silhouette_score')
plt.grid(True)
# 用k的取值作为横轴刻度
plt.xticks(Ks)
plt.show()
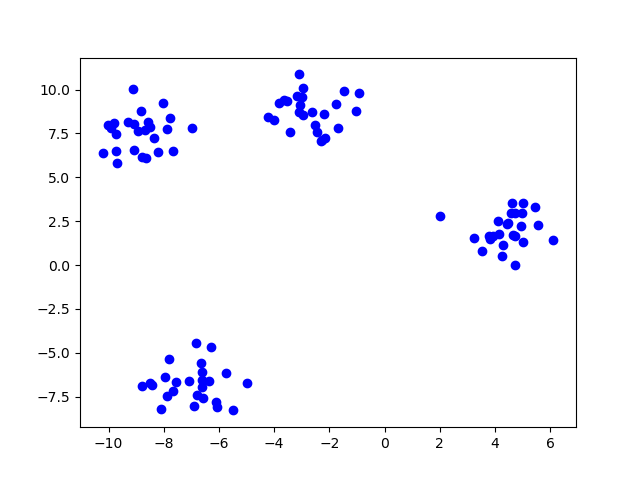
样本点图
绘制的整体轮廓系数与k的关系曲线
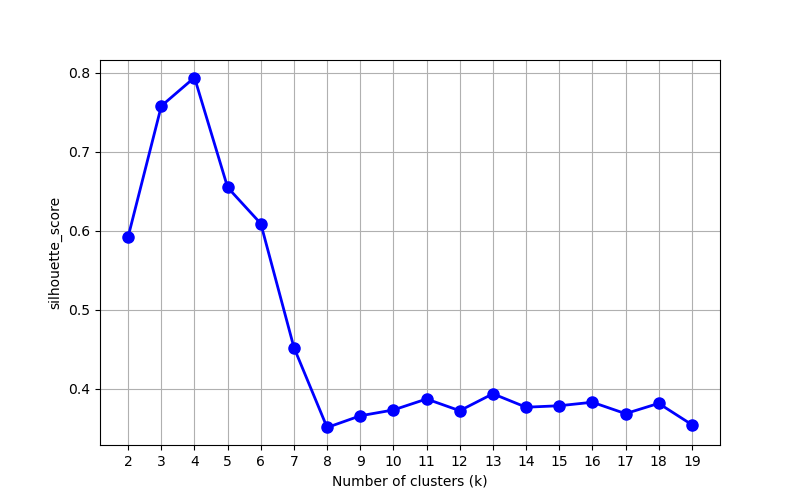
可以看到当k=4时,整体轮廓系数最大,也就是说,分成4簇是最优的
Calinski-Harabasz 指数/方差比准则
核心思想:簇间离散度越大越好,簇内离散度越小越好
直观上理解就是每个簇内部抱得很紧,外部与其他簇保持距离,形成很鲜明的聚类
CH=B(k)W(k)×N−kk−1B(k):簇间离散度B(k)=∑i=1kni∣∣μi−μ∣∣2ni:第i个簇的样本数μi:第i个簇的中心μ:整个数据集的均值向量W(k):簇内离散度W(k)=WCSS=∑i=1k∑x∈c(i)∣∣x−μi∣∣2N:样本总数k:簇的数量 CH=\frac{B(k)}{W(k)}\times\frac{N-k}{k-1}\\ B(k):簇间离散度\\ B(k)= \sum_{i=1}^{k}n_i||\mu_i-\mu||^2\\ n_i:第i个簇的样本数\\ \mu_i:第i个簇的中心\\ \mu:整个数据集的均值向量\\ W(k):簇内离散度\\ W(k)=WCSS = \sum_{i=1}^{k}\sum_{x \in c^{(i)}}||x-\mu_i||^2\\ N:样本总数\\ k:簇的数量\\ CH=W(k)B(k)×k−1N−kB(k):簇间离散度B(k)=i=1∑kni∣∣μi−μ∣∣2ni:第i个簇的样本数μi:第i个簇的中心μ:整个数据集的均值向量W(k):簇内离散度W(k)=WCSS=i=1∑kx∈c(i)∑∣∣x−μi∣∣2N:样本总数k:簇的数量
sklearn实现
from sklearn.metrics import calinski_harabasz_score as CH
选择最优k并绘制CH与k的关系曲线
def get_best_K_by_CH(Ks):
X, y = make_blobs(
n_samples=100,
centers=4,
random_state=42,
)
plt.scatter(X[:, 0], X[:, 1], c='b')
scores = []
max_score = -1
best_k = 0
for k in Ks:
kmeans = KMeans(n_clusters=k)
kmeans.fit(X)
labels = kmeans.labels_
score = CH(X, labels)
print(score)
if score > max_score:
max_score = score
best_k = k
scores.append(score)
plt.figure(figsize=(8, 5))
plt.plot(Ks, scores, 'bo-', linewidth=2, markersize=8)
plt.xlabel('Number of clusters (k)')
plt.ylabel('CH')
plt.grid(True)
# 用k的取值作为横轴刻度
plt.xticks(Ks)
plt.show()
return best_k
得到的CH与k的关系曲线
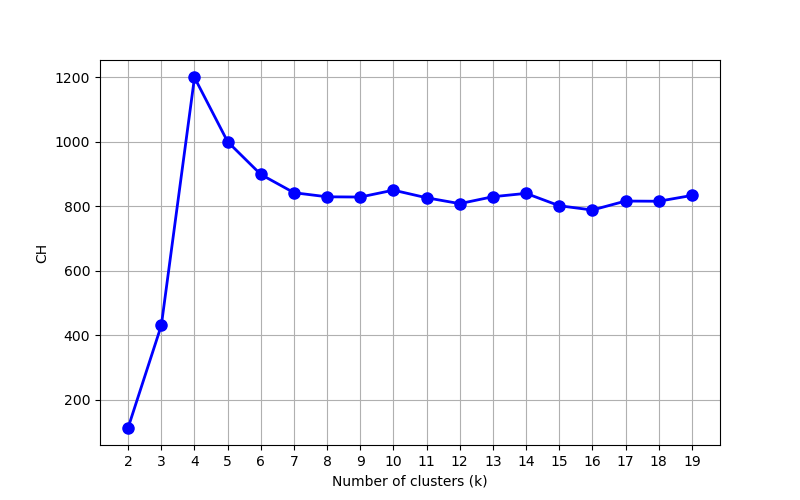
最优k为4