背景
糖尿病数据集,442个样本,10个特征(年龄、血压等),目标为疾病进展值
该数据集为回归问题,需要使用回归分析方法进行分析
数据集速览
import pandas as pd
from sklearn.datasets import load_diabetes
diabetes = load_diabetes()
# 将数据转换为 Pandas DataFrame 以便更好地查看
# features(特征)
df_features = pd.DataFrame(diabetes.data, columns=diabetes.feature_names)
# target(目标变量)
df_target = pd.DataFrame(diabetes.target, columns=['target'])
# 合并特征和目标变量为一个 DataFrame
df_diabetes = pd.concat([df_features, df_target], axis=1)
print("--- 数据集简要概览 ---")
print(f"数据形状 (行, 列): {df_diabetes.shape}")
print("\n数据集特征名称:")
print(diabetes.feature_names)
print("\n目标变量描述:")
print(diabetes.DESCR.split('\n')[-3]) # 提取目标变量的简要描述
print("\n--- 数据前5行预览 ---")
print(df_diabetes.head())
print("\n--- 数据信息 (类型和非空值) ---")
print(df_diabetes.info())
--- 数据集简要概览 ---
数据形状 (行, 列): (442, 11)
数据集特征名称:
['age', 'sex', 'bmi', 'bp', 's1', 's2', 's3', 's4', 's5', 's6']
目标变量描述:
Bradley Efron, Trevor Hastie, Iain Johnstone and Robert Tibshirani (2004) "Least Angle Regression," Annals of Statistics (with discussion), 407-499.
--- 数据前5行预览 ---
age sex bmi bp s1 s2 s3 \
0 0.038076 0.050680 0.061696 0.021872 -0.044223 -0.034821 -0.043401
1 -0.001882 -0.044642 -0.051474 -0.026328 -0.008449 -0.019163 0.074412
2 0.085299 0.050680 0.044451 -0.005670 -0.045599 -0.034194 -0.032356
3 -0.089063 -0.044642 -0.011595 -0.036656 0.012191 0.024991 -0.036038
4 0.005383 -0.044642 -0.036385 0.021872 0.003935 0.015596 0.008142
s4 s5 s6 target
0 -0.002592 0.019907 -0.017646 151.0
1 -0.039493 -0.068332 -0.092204 75.0
2 -0.002592 0.002861 -0.025930 141.0
3 0.034309 0.022688 -0.009362 206.0
4 -0.002592 -0.031988 -0.046641 135.0
--- 数据信息 (类型和非空值) ---
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 442 entries, 0 to 441
Data columns (total 11 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 age 442 non-null float64
1 sex 442 non-null float64
2 bmi 442 non-null float64
3 bp 442 non-null float64
4 s1 442 non-null float64
5 s2 442 non-null float64
6 s3 442 non-null float64
7 s4 442 non-null float64
8 s5 442 non-null float64
9 s6 442 non-null float64
10 target 442 non-null float64
dtypes: float64(11)
memory usage: 38.1 KB
None
这份数据集共有 10 个特征
它们都是经过预处理和标准化的浮点数值,具体含义如下:
1. age: 年龄(years)
2. sex: 性别
3. bmi: 身体质量指数(body mass index)
4. bp: 平均血压(average blood pressure)
5. s1: T-细胞(T-cells)
6. s2: 低密度脂蛋白(LDL)
7. s3: 高密度脂蛋白(HDL)
8. s4: 甲状腺刺激素(TSH)
9. s5: 血糖(blood sugar)
10. s6: 淋巴细胞(lymphocytes)
分析方法
- 线性回归
- K近邻(KNN)
- 决策树
- 支持向量机
- 随机森林
- 多层感知器(MLP)
- Lasso 回归
- Ridge 回归
- 梯度提升回归
- AdaBoost 回归
- Huber 回归
分析步骤
- 加载数据集
- 拆分训练集、测试集
- 数据预处理(标准化)
- 选择模型
- 模型训练(拟合)
- 测试模型效果
- 评估模型
分析结果
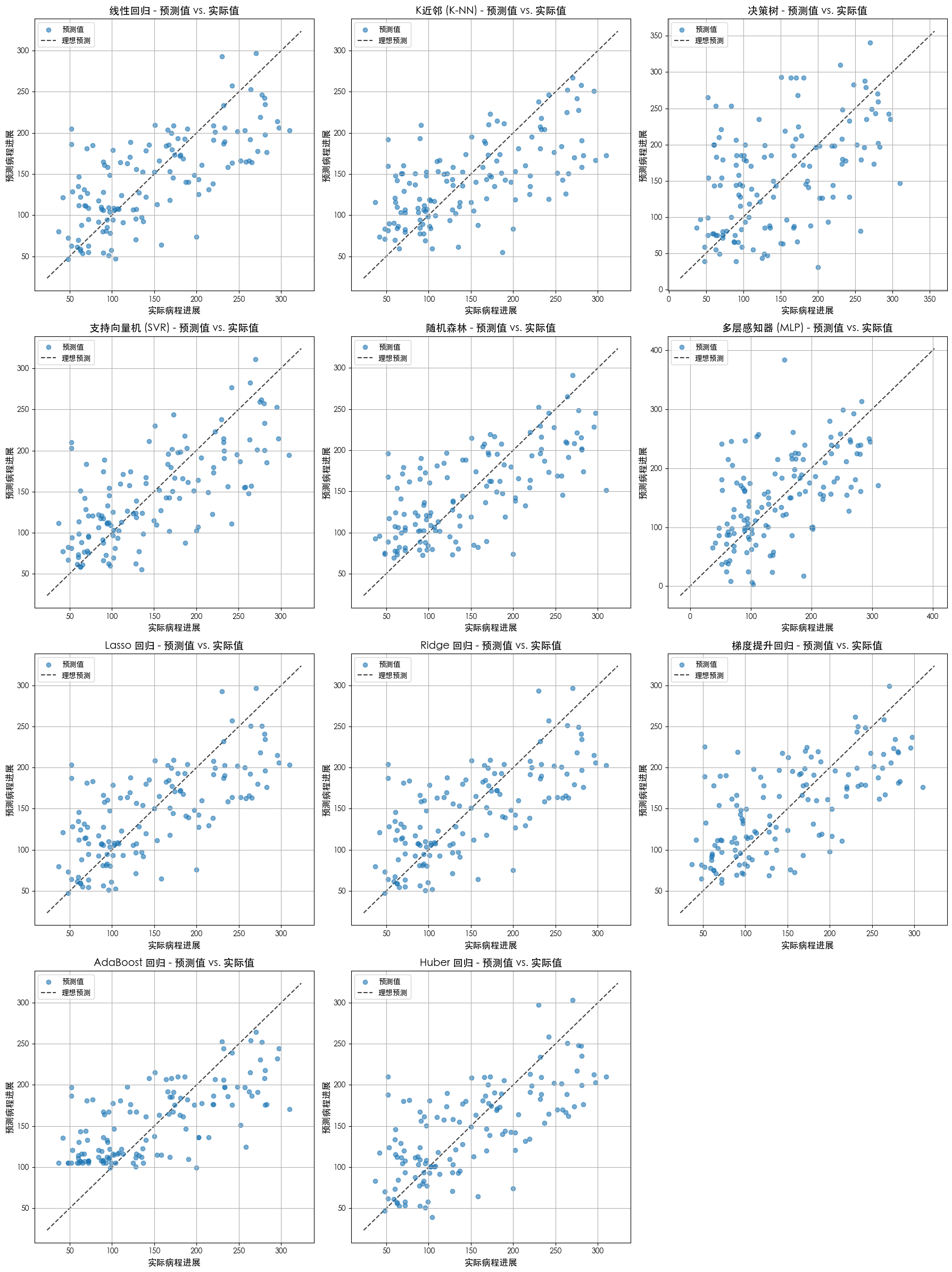
不同模型的预测值 vs 真实值效果对比
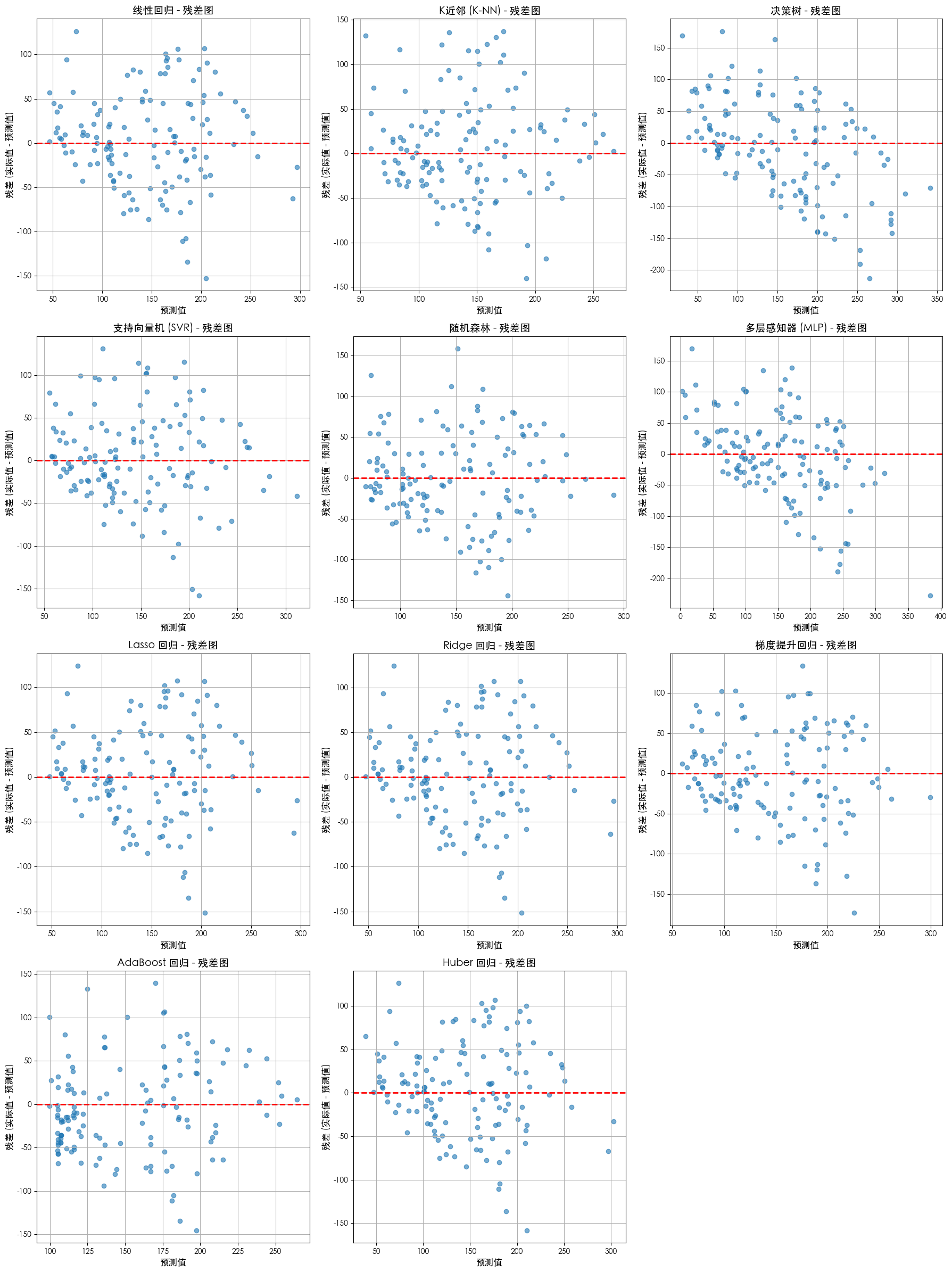
残差图
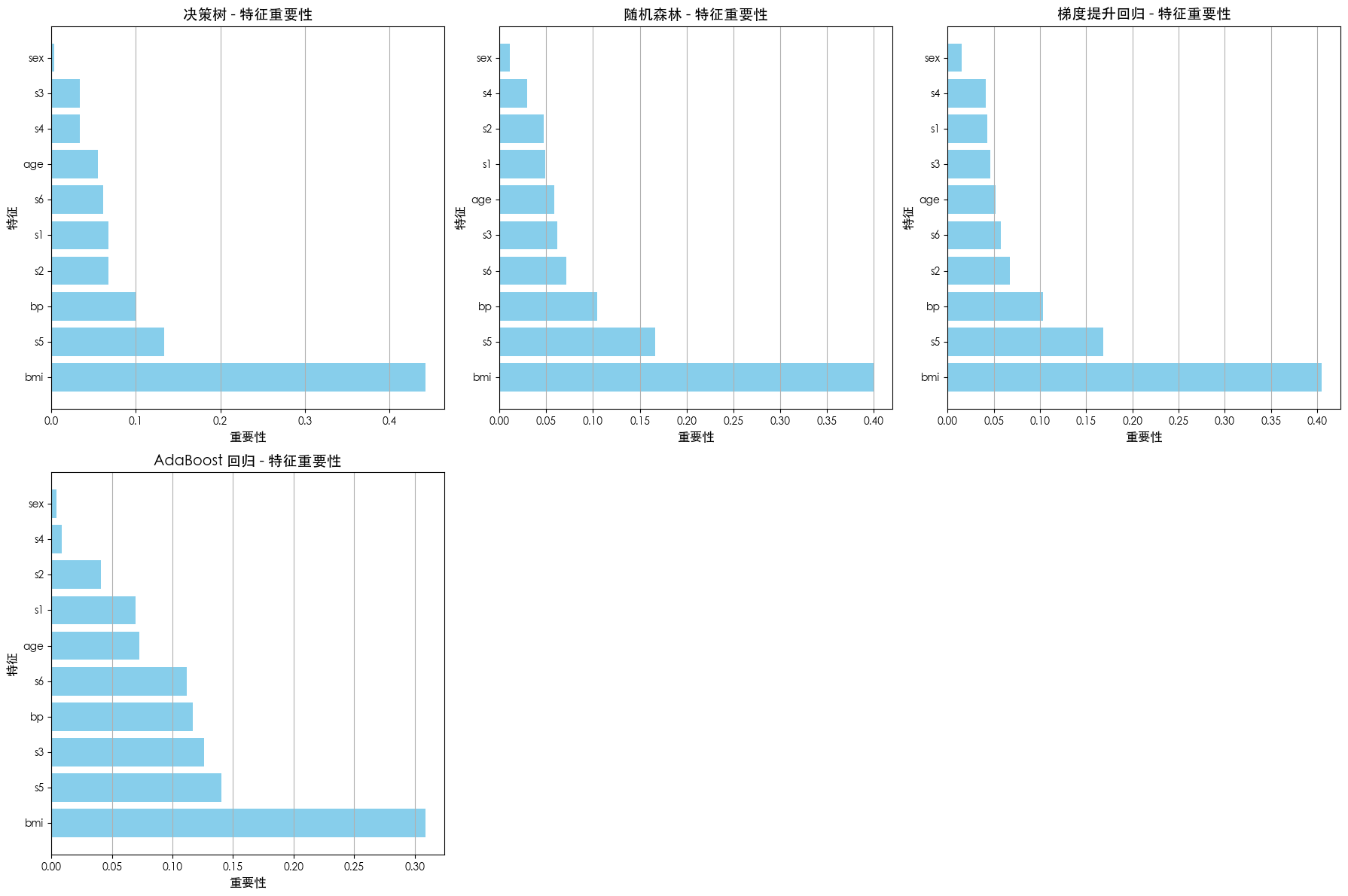
特征重要性
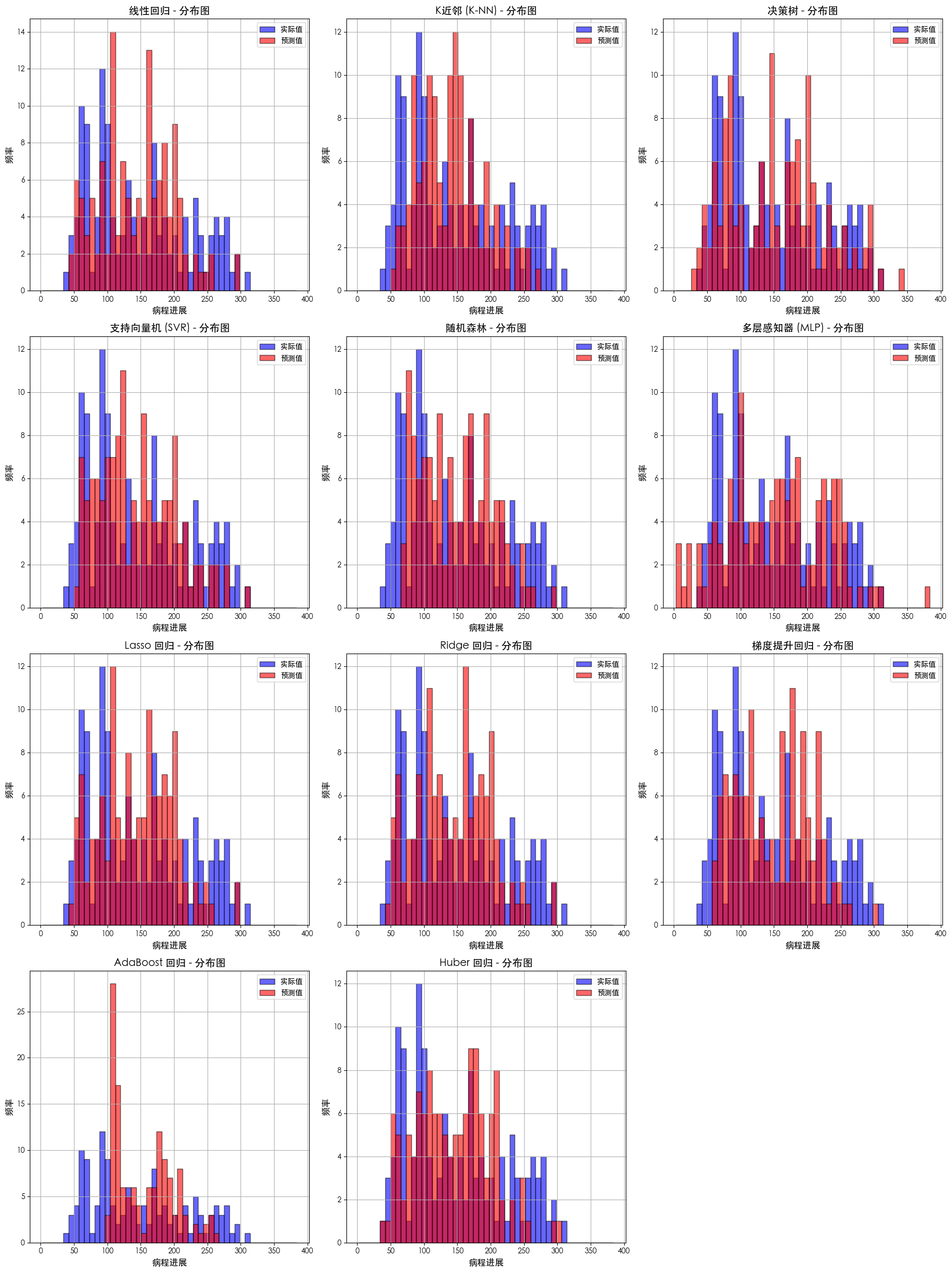
预测值分布
不同模型评估明细
--- 数据集概览 ---
数据形状: (442, 10)
特征名称: ['age', 'sex', 'bmi', 'bp', 's1', 's2', 's3', 's4', 's5', 's6']
目标变量名称: 糖尿病病程进展
--- 数据划分结果 ---
训练集形状: (309, 10)
测试集形状: (133, 10)
--- 正在对数据进行标准化处理 ---
--- 模型训练与评估 ---
--- 正在训练 线性回归 模型 ---
线性回归 模型的均方误差 (MSE): 2821.7510
线性回归 模型的 R^2 得分: 0.4773
--- 正在训练 K近邻 (K-NN) 模型 ---
K近邻 (K-NN) 模型的均方误差 (MSE): 3277.7368
K近邻 (K-NN) 模型的 R^2 得分: 0.3928
--- 正在训练 决策树 模型 ---
决策树 模型的均方误差 (MSE): 5686.6015
决策树 模型的 R^2 得分: -0.0534
--- 正在训练 支持向量机 (SVR) 模型 ---
支持向量机 (SVR) 模型的均方误差 (MSE): 2822.7007
支持向量机 (SVR) 模型的 R^2 得分: 0.4771
--- 正在训练 随机森林 模型 ---
随机森林 模型的均方误差 (MSE): 2859.3911
随机森林 模型的 R^2 得分: 0.4703
--- 正在训练 多层感知器 (MLP) 模型 ---
/opt/anaconda3/lib/python3.13/site-packages/sklearn/neural_network/_multilayer_perceptron.py:691: ConvergenceWarning: Stochastic Optimizer: Maximum iterations (20000) reached and the optimization hasn't converged yet.
warnings.warn(
多层感知器 (MLP) 模型的均方误差 (MSE): 4791.4414
多层感知器 (MLP) 模型的 R^2 得分: 0.1124
--- 正在训练 Lasso 回归 模型 ---
Lasso 回归 模型的均方误差 (MSE): 2817.0876
Lasso 回归 模型的 R^2 得分: 0.4782
--- 正在训练 Ridge 回归 模型 ---
Ridge 回归 模型的均方误差 (MSE): 2819.9820
Ridge 回归 模型的 R^2 得分: 0.4776
--- 正在训练 梯度提升回归 模型 ---
梯度提升回归 模型的均方误差 (MSE): 3083.3332
梯度提升回归 模型的 R^2 得分: 0.4288
--- 正在训练 AdaBoost 回归 模型 ---
AdaBoost 回归 模型的均方误差 (MSE): 2874.6206
AdaBoost 回归 模型的 R^2 得分: 0.4675
--- 正在训练 Huber 回归 模型 ---
Huber 回归 模型的均方误差 (MSE): 2828.7736
Huber 回归 模型的 R^2 得分: 0.4760
所有模型的分析已完成。
代码
from sklearn.datasets import load_diabetes
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.neighbors import KNeighborsRegressor
from sklearn.tree import DecisionTreeRegressor
from sklearn.svm import SVR
from sklearn.linear_model import LinearRegression, Lasso, Ridge, HuberRegressor
from sklearn.ensemble import RandomForestRegressor, GradientBoostingRegressor, AdaBoostRegressor
from sklearn.neural_network import MLPRegressor
from sklearn.metrics import mean_squared_error, r2_score
import numpy as np
import matplotlib.pyplot as plt
# 设置 Matplotlib 字体以正确显示中文
plt.rcParams['font.sans-serif'] = ['SimHei', 'WenQuanYi Zen Hei', 'STHeiti', 'Arial Unicode MS']
plt.rcParams['axes.unicode_minus'] = False
def perform_diabetes_analysis():
"""
使用 scikit-learn 对糖尿病数据集进行全面的分析。
该函数包含数据加载、预处理、模型训练、评估和结果可视化。
"""
print("--- 正在加载糖尿病数据集 ---")
diabetes = load_diabetes()
X = diabetes.data
y = diabetes.target
feature_names = list(diabetes.feature_names)
print("\n--- 数据集概览 ---")
print(f"数据形状: {X.shape}")
print(f"特征名称: {feature_names}")
print(f"目标变量名称: {'糖尿病病程进展'}")
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=42)
print("\n--- 数据划分结果 ---")
print(f"训练集形状: {X_train.shape}")
print(f"测试集形状: {X_test.shape}")
print("\n--- 正在对数据进行标准化处理 ---")
scaler = StandardScaler()
X_train_scaled = scaler.fit_transform(X_train)
X_test_scaled = scaler.transform(X_test)
models = {
"线性回归": LinearRegression(),
"K近邻 (K-NN)": KNeighborsRegressor(n_neighbors=5),
"决策树": DecisionTreeRegressor(random_state=42),
"支持向量机 (SVR)": SVR(kernel='rbf', C=100),
"随机森林": RandomForestRegressor(random_state=42),
"多层感知器 (MLP)": MLPRegressor(random_state=42, max_iter=20000),
"Lasso 回归": Lasso(alpha=0.1, max_iter=10000),
"Ridge 回归": Ridge(alpha=1.0, max_iter=10000),
"梯度提升回归": GradientBoostingRegressor(random_state=42),
"AdaBoost 回归": AdaBoostRegressor(random_state=42),
"Huber 回归": HuberRegressor(max_iter=10000)
}
predictions = {}
feature_importances = {}
print("\n--- 模型训练与评估 ---")
for name, model in models.items():
print(f"\n--- 正在训练 {name} 模型 ---")
model.fit(X_train_scaled, y_train)
y_pred = model.predict(X_test_scaled)
mse = mean_squared_error(y_test, y_pred)
r2 = r2_score(y_test, y_pred)
print(f"{name} 模型的均方误差 (MSE): {mse:.4f}")
print(f"{name} 模型的 R^2 得分: {r2:.4f}")
predictions[name] = y_pred
if hasattr(model, 'feature_importances_'):
feature_importances[name] = model.feature_importances_
print("\n所有模型的分析已完成。")
# --- 预测值 vs. 实际值可视化 ---
print("\n--- 预测值 vs. 实际值散点图 ---")
num_models = len(models)
cols = 3
rows = (num_models + cols - 1) // cols
fig, axes = plt.subplots(rows, cols, figsize=(18, 6 * rows))
axes = axes.flatten()
for i, name in enumerate(models.keys()):
ax = axes[i]
y_pred = predictions[name]
ax.scatter(y_test, y_pred, alpha=0.6, label='预测值')
lims = [
np.min([ax.get_xlim(), ax.get_ylim()]),
np.max([ax.get_xlim(), ax.get_ylim()]),
]
ax.plot(lims, lims, 'k--', alpha=0.75, zorder=0, label='理想预测')
ax.set_title(f'{name} - 预测值 vs. 实际值', fontsize=14)
ax.set_xlabel('实际病程进展', fontsize=12)
ax.set_ylabel('预测病程进展', fontsize=12)
ax.legend()
ax.grid(True)
for j in range(num_models, len(axes)):
axes[j].axis('off')
plt.tight_layout()
plt.show()
# --- 残差图可视化 ---
print("\n--- 残差图可视化 ---")
fig, axes = plt.subplots(rows, cols, figsize=(18, 6 * rows))
axes = axes.flatten()
for i, name in enumerate(models.keys()):
ax = axes[i]
y_pred = predictions[name]
residuals = y_test - y_pred
ax.scatter(y_pred, residuals, alpha=0.6)
ax.axhline(y=0, color='r', linestyle='--', linewidth=2)
ax.set_title(f'{name} - 残差图', fontsize=14)
ax.set_xlabel('预测值', fontsize=12)
ax.set_ylabel('残差 (实际值 - 预测值)', fontsize=12)
ax.grid(True)
for j in range(num_models, len(axes)):
axes[j].axis('off')
plt.tight_layout()
plt.show()
# --- 特征重要性可视化 (仅限树模型) ---
if feature_importances:
print("\n--- 特征重要性可视化 ---")
num_tree_models = len(feature_importances)
rows_tree = (num_tree_models + cols - 1) // cols
fig, axes = plt.subplots(rows_tree, cols, figsize=(18, 6 * rows_tree))
axes = axes.flatten()
for i, (name, importances) in enumerate(feature_importances.items()):
ax = axes[i]
sorted_indices = np.argsort(importances)[::-1]
sorted_importances = importances[sorted_indices]
sorted_feature_names = [feature_names[j] for j in sorted_indices]
ax.barh(sorted_feature_names, sorted_importances, color='skyblue')
ax.set_title(f'{name} - 特征重要性', fontsize=14)
ax.set_xlabel('重要性', fontsize=12)
ax.set_ylabel('特征', fontsize=12)
ax.grid(axis='x')
for j in range(num_tree_models, len(axes)):
axes[j].axis('off')
plt.tight_layout()
plt.show()
# --- 预测值分布图可视化 ---
print("\n--- 预测值分布图 ---")
fig, axes = plt.subplots(rows, cols, figsize=(18, 6 * rows))
axes = axes.flatten()
all_values = np.concatenate([y_test] + list(predictions.values()))
bins = np.linspace(min(all_values), max(all_values), 50)
for i, name in enumerate(models.keys()):
ax = axes[i]
y_pred = predictions[name]
ax.hist(y_test, bins=bins, alpha=0.6, label='实际值', color='b', edgecolor='k')
ax.hist(y_pred, bins=bins, alpha=0.6, label='预测值', color='r', edgecolor='k')
ax.set_title(f'{name} - 分布图', fontsize=14)
ax.set_xlabel('病程进展', fontsize=12)
ax.set_ylabel('频率', fontsize=12)
ax.legend()
ax.grid(True)
for j in range(num_models, len(axes)):
axes[j].axis('off')
plt.tight_layout()
plt.show()
if __name__ == "__main__":
perform_diabetes_analysis()