单细胞RNA测序揭示了肺腺癌中不同的肿瘤微环境模式

摘要
免疫肿瘤学的最新进展表明,不仅癌细胞,肿瘤微环境也能指导精准医疗。由于肿瘤微环境中的细胞种类繁多,即便数量稀少也可能具有重要意义,因此对其进行全面深入的表征颇具挑战性。为了识别具有临床意义的微环境和癌症特征,我们对10例人类肺腺癌组织和10例正常对照组织进行了单细胞RNA测序。我们的分析揭示了异质性的癌细胞转录组,反映了组织学分级和致癌通路的活性,以及两种截然不同的微环境模式。免疫激活的CP²E微环境由癌相关肌成纤维细胞、促炎性单核细胞衍生巨噬细胞、浆细胞样树突状细胞和耗竭的CD8+T细胞组成,预后不良。相比之下,惰性的N³MC微环境则以正常样肌成纤维细胞、非炎性单核细胞衍生巨噬细胞、自然杀伤细胞、髓样树突状细胞和常规 T细胞为特征,与良好的预后相关。在单细胞图谱中鉴定出的微环境标记基因和特征在肿瘤总体图谱中具有预后价值。总之,对肺腺癌进行单细胞RNA测序可基于微环境提供额外的预后信息,并可能有助于预测治疗反应以及揭示未来治疗手段的潜在靶细胞群。
数据收集
12 Lung tumor patients
软件使用
软件包信息
软件包名称 |
应用 |
链接 |
关键功能说明 |
|---|---|---|---|
| CellPhoneDB | 细胞互作分析 |
cellphonedb.org |
基于配体-受体对的细胞间通讯预测,支持单细胞RNA-seq数据 |
| Seurat v3 | 单细胞数据分析 |
satijalab.org/seurat |
单细胞RNA-seq聚类、差异表达分析、数据可视化 |
| ImageJ | 生物图像分析 |
imagej.nih.gov/ij/ |
多模态图像处理、定量分析(如荧光强度、细胞计数) |
| Sequence Pilot (v4.4.0, JSI) | NGS测序设计 |
jsi-medisys.com |
引物/探针设计、测序实验自动化规划 |
| Ion Reporter (v5.12, ThermoFisher) | NGS数据分析 |
thermofisher.com/ionreporter |
变异检测、注释及临床报告生成(兼容Ion Torrent数据) |
| R (Bioconductor等包) | 统计与生信分析 |
r-project.org |
通用数据分析、支持单细胞/转录组/基因组分析(如DESeq2、limma) |
统计方法信息
Welch’s t-test
用途:比较两个独立样本的均值,适用于方差不等或样本量不等的情况
假设:
-
数据连续且独立
近似正态分布(但对方差齐性无要求)
R函数:
t.test(..., var.equal = FALSE)
Mann–Whitney U test
别名:Wilcoxon rank-sum test
用途:非参数检验,比较两个独立样本的中位数(或分布形状)
适用条件:
-
数据至少为序数尺度
不满足正态假设时使用
R函数:
wilcox.test()
Shapiro–Wilk test
用途:检验数据是否来自正态分布
假设:
-
H₀:样本来自正态分布
H₁:样本不来自正态分布
注意:对小样本(n < 50)更敏感
R函数:
shapiro.test()
实验验证方法
Collection of Tissue Specimens(组织样本收集)
目的
获取高质量的新鲜或冷冻组织样本,用于单细胞测序和后续验证实验。
方法
样本来源:
-
手术切除、活检或尸检组织
匹配癌组织及邻近正常组织(如适用)
样本处理:
-
立即置于预冷PBS或保存介质(如RPMI-1640)
部分组织速冻于液氮(-80°C保存)
部分组织固定于4%多聚甲醛(用于H&E/免疫染色)
质量控制:
-
病理评估(确保肿瘤含量>70%)
RNA完整性检测(RIN > 7)
Tissue Dissociation and Single-Cell Isolation(组织解离与单细胞分离)
目的
获得高活性、低碎片率的单细胞悬液。
方法
机械解离:
-
使用GentleMACS或手术刀切碎组织
酶消化:
-
根据组织类型选择酶(如胶原酶IV/DNase I)
37°C孵育优化时间(避免过度消化)
细胞筛选:
-
70μm滤网去除团块
红细胞裂解(ACK缓冲液)
死细胞去除(如Dead Cell Removal Kit)
质检:
-
台盼蓝染色评估活性(>85%)
流式检测细胞浓度(目标500-1,200 cells/μL)
Single-Cell RNA Sequencing(单细胞RNA测序)
目的
生成高质量单细胞转录组数据。
方法
文库构建:
-
10x Genomics Chromium平台(3’/5’端或全转录组)
或Smart-seq2(全长转录本)
测序参数:
-
目标测序深度:50,000 reads/cell(10x)
双端测序(PE150)
对照设置:
-
加入spike-in RNA(如ERCC)
空白孔对照检测环境RNA污染
H&E and Immunostaining(H&E与免疫组化染色)
目的
验证组织病理特征及目标蛋白表达。
方法
H&E染色:
-
福尔马林固定石蜡包埋(FFPE)切片
标准苏木精-伊红染色流程
免疫染色:
-
抗原修复(柠檬酸缓冲液热诱导)
一抗孵育(如PD-L1、CK7等标志物)
DAB显色或荧光标记(IF)
分析:
-
病理医师盲法评分
QuPath软件定量染色强度
Panel Sequencing(靶向Panel测序)
目的
验证关键基因突变或表达变异。
方法
Panel设计:
-
针对癌相关基因(如NSCLC panel涵盖EGFR/KRAS/TP53)
建库:
-
DNA/RNA提取(Qiagen Kit)
杂交捕获(如Agilent SureSelect)
测序:
-
Illumina NovaSeq(目标深度:DNA 500×,RNA 100×)
分析:
-
GATK标准流程检测SNV/Indel
RNA-Seq比对(STAR)及融合基因检测(Arriba)
研究成果
肺腺癌的单细胞RNA测序
A 工作流程示意图。
B,C 基于单细胞转录组中前15个主成分的UMAP图经过筛选后,按(B)组织类型或(C)患者进行颜色编码。
D 临床特征概述、与临床相关的致癌突变和基因融合情况;每个患者主要细胞类型的数量统计以及所有单细胞转录组的UMAP图。
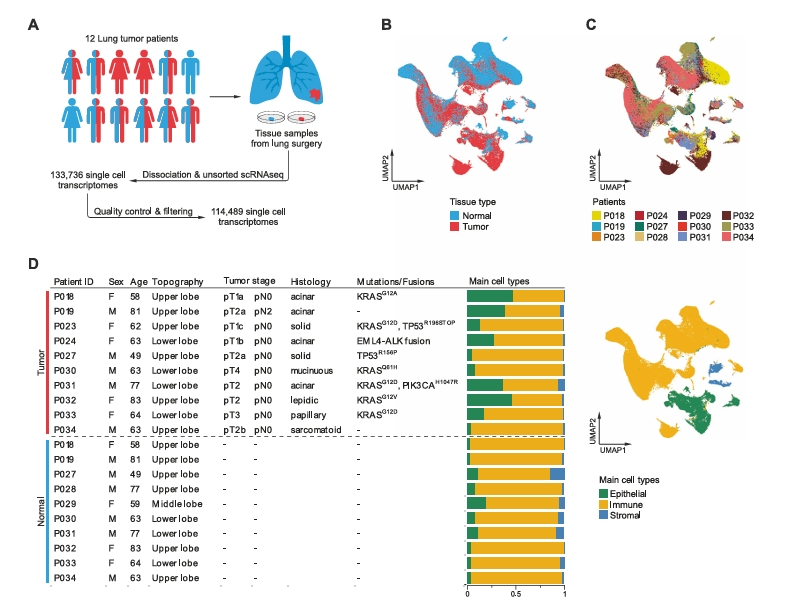
肺腺癌肿瘤上皮细胞之间的肿瘤间异质性
A 基于所有上皮单细胞转录组的前20个主成分构建的UMAP图。
B 选定的正常上皮细胞类型标记基因的平均基因表达情况。
C 按患者分组的肿瘤上皮细胞中差异表达的基因,每个患者的最大前10个基因显示情况
D 由选定的差异表达基因编码的蛋白质的免疫组织化学染色。
E 按患者分组的肿瘤上皮细胞的平均通路活性得分。
F 组织学亚型的分布,(G)正常上皮细胞类型基因签名的平均模块得分,以及(H)按主成分1(PC1)排序的肿瘤上皮细胞的平均通路活性得分。
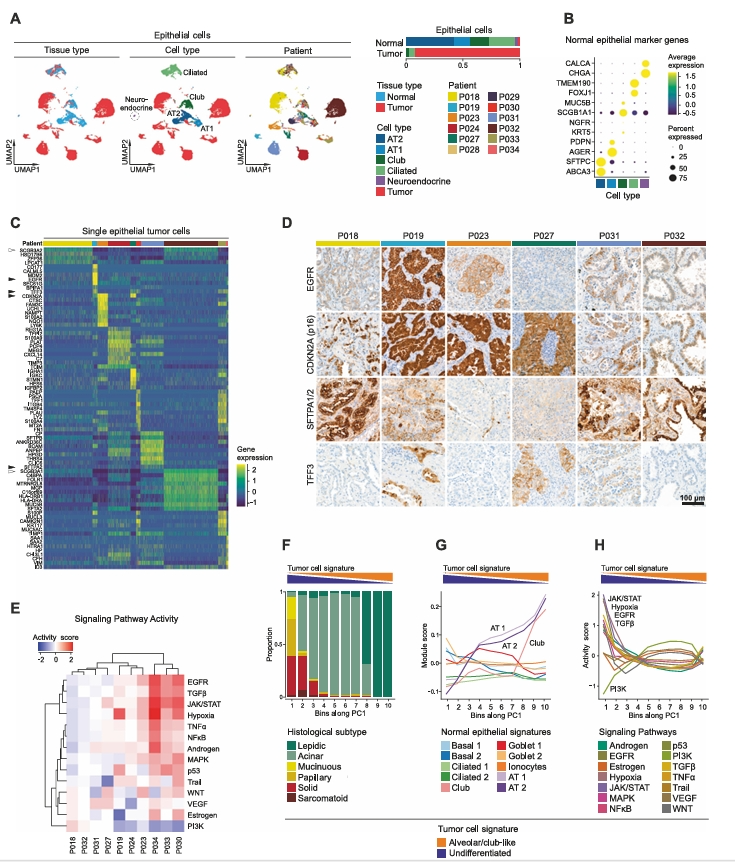
肺腺癌肿瘤微环境的组成
A 基于所有间质单细胞转录组中前20个主成分的UMAP分析,以颜色编码表示细胞簇;并按组织类型对内皮细胞和成纤维细胞/肌肉细胞簇的相对数量进行定量。
B 选定的间质细胞簇标记基因的平均基因表达量。
C 成纤维细胞/肌肉细胞簇的差异表达基因,每个细胞簇的最大显著程度。
D 各种成纤维细胞/肌肉细胞簇的平均通路活性得分。
E 1和2型肌成纤维细胞簇的相对数量的相关性。
F 作为肌成纤维细胞簇2标记物的CTHRC1免疫组织化学染色,CTHRC1+细胞覆盖的间质区域比例的定量。
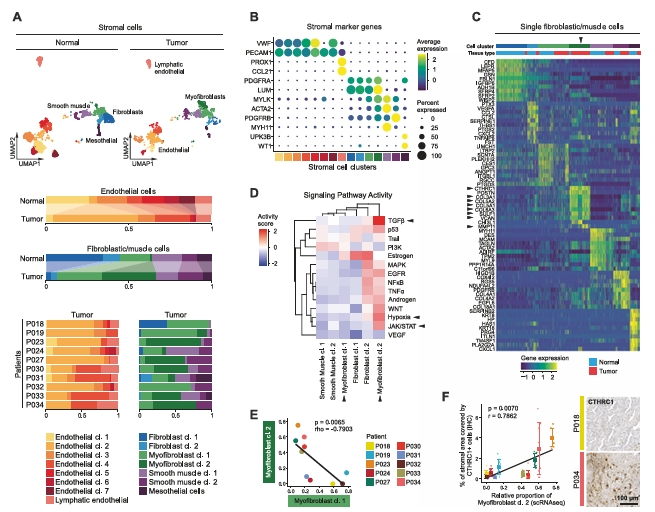
肺腺癌免疫微环境的构成
A 基于所有免疫单细胞转录组中前20个主成分并按组织类型划分、以颜色编码表示细胞簇的UMAP图。
B 选定的免疫细胞簇标记基因的平均基因表达量。
C 与炎症相关的基因签名以及不同巨噬细胞簇的M1/M2极化相关的模块得分。
D 选定的髓系免疫细胞簇的相对数量。
E CXCL9和CD123的免疫组织化学染色作为单核细胞衍生巨噬细胞簇2和浆细胞样树突状细胞的标志物。
F 基因模块得分。与不同CD8+T细胞簇的细胞毒性及耗竭相关的特征标记。
G 选定的淋巴细胞和髓系免疫细胞簇的相对数量的相关性。
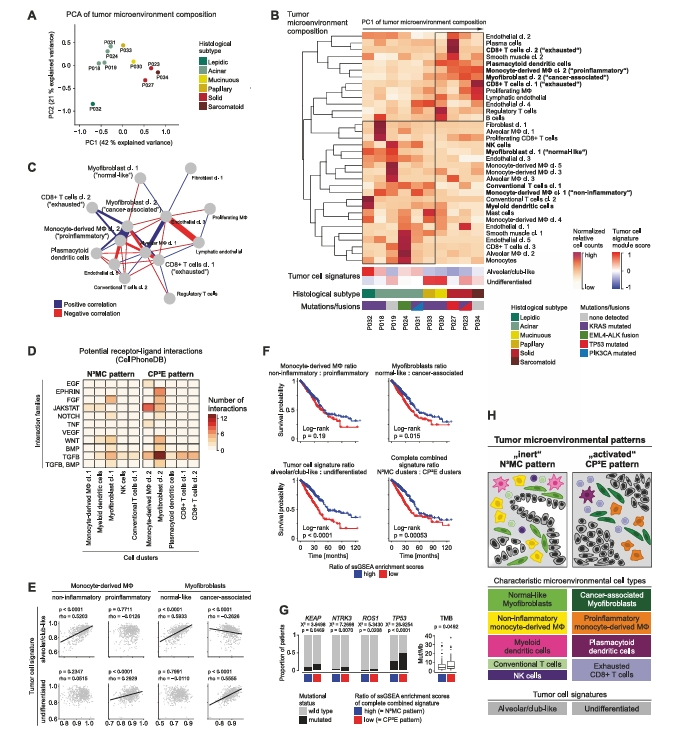
肺腺癌的肿瘤微环境模式
A 基于间质细胞和免疫细胞簇的比例进行的主成分分析。
B 标准化间质细胞和免疫细胞簇的比例、肿瘤细胞特征的平均模块得分、每位患者的组织学亚型和突变状态。
C 间质细胞和免疫细胞簇比例的相关性,展示相关网络图中连接最紧密的部分。
D 由微环境细胞簇到N³MC或CP²E模式肿瘤细胞的潜在旁分泌相互作用的数量。
E 基于选定的微环境细胞簇和肿瘤细胞特征的标记基因的ssGSEA富集分数的相关性。
F Kaplan-Meier整体生存曲线。
G 患者中具有致癌突变和肿瘤突变负荷的比例。
H 肺腺癌亚型的示意图。
Reference
Bischoff P, et al. Single-cell RNA sequencing reveals distinct tumor microenvironmental patterns in lung adenocarcinoma. Oncogene. 2021 Dec;40(50):6748-6758. doi: 10.1038/s41388-021-02054-3. Epub 2021 Oct 18. PMID: 34663877; PMCID: PMC8677623.
