之前我们推出过单细胞差异丰度分析的R包miloR(miloR:单细胞差异丰度分析)。这个方法也有python版的实现,github官网: https://github.com/emdann/milopy, 如果你的单细胞流程是基于python的,那么就无需转化了,以免转化过程出问题,可以直接使用python版本进行分析。需要注意的是,milopy这个包已经被整合到pertpy这里了,之前的可能不再维护,所以我们直接介绍pertpy教程。总体而言,步骤和miloR差不多!
其实pertpy我们在上节已经介绍过了(scCODA:单细胞差异成分分析):
最新的论文在预印本:https://www.biorxiv.org/content/10.1101/2024.08.04.606516v1
所以这里我们安装pertpy github: https://github.com/scverse/pertpy
安装:
#终端
conda create -n pertpy python=3.10
conda activate pertpy
#安装包
pip install pertpy -i https://pypi.tuna.tsinghua.edu.cn/simple
import matplotlib
import matplotlib.pyplot as plt
import pertpy as pt
import scanpy as sc
import numpy as np
sc.settings.verbosity = 3加载数据(scanpy object, h5ad数据):
#load data
adata = sc.read_h5ad('./miloData.h5ad')
adata
#AnnData object with n_obs × n_vars = 17270 × 15272
# obs: 'orig.ident', 'nCount_RNA', 'nFeature_RNA', 'percent.mito', 'group', 'RNA_snn_res.0.4', 'seurat_clusters', 'celltype', 'RNA_snn_res.0.2', 'RNA_snn_res.0.3'
# var: 'vst.mean', 'vst.variance', 'vst.variance.expected', 'vst.variance.standardized', 'vst.variable'
# obsm: 'X_pca', 'X_tsne', 'X_umap'
#展示数据,非必要运行
sc.pl.umap(adata, color=["celltype"], legend_loc="on data")
sc.pl.umap(adata, color=["orig.ident",'group'], wspace=0.3, size=8)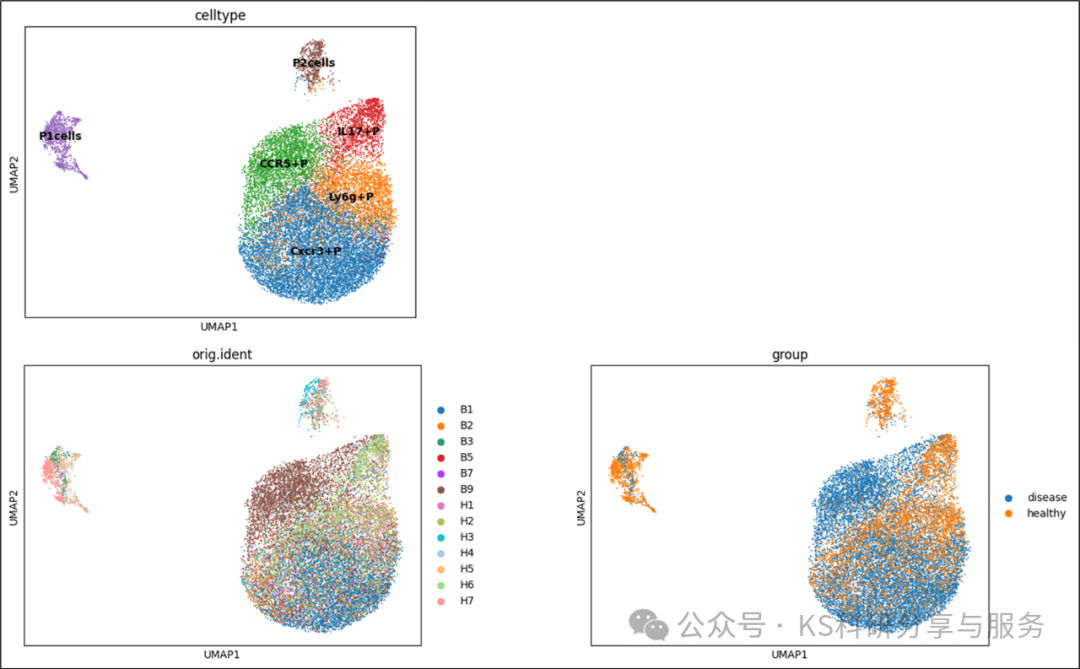
分析准备:初始化Milo对象,创建一个MuData对象(这是pertpy这个库的特点,之前讲说的scCODA也是如此),该对象将存储基因表达矩阵(rna modalities:也就是我们原始的adata)和用于差异丰度分析的细胞计数矩阵(milo modalities)。
## Initialize object for Milo analysis
milo = pt.tl.Milo()
mdata = milo.load(adata)
mdata
MuData object with n_obs × n_vars = 17270 × 15272
2 modalities
rna: 17270 x 15272
obs: 'orig.ident', 'nCount_RNA', 'nFeature_RNA', 'percent.mito', 'group', 'RNA_snn_res.0.4', 'seurat_clusters', 'celltype', 'RNA_snn_res.0.2', 'RNA_snn_res.0.3'
var: 'vst.mean', 'vst.variance', 'vst.variance.expected', 'vst.variance.standardized', 'vst.variable'
uns: 'celltype_colors', 'orig.ident_colors', 'dendrogram_celltype'
obsm: 'X_pca', 'X_tsne', 'X_umap'
milo: 0 x 0
Build KNN graph:
sc.pp.neighbors(mdata["rna"],n_neighbors=50)
Construct neighbourhoods:
milo.make_nhoods(mdata["rna"], prop=0.2,seed=1234)
#input data是MuData对象中的‘rna',原始的经过knn的adata
#prop是用于搜索邻域指数的样本细胞比例。一般0.1-0.2
#seed:随机种子
#可以根据分布调整前面的参数。
nhood_size = np.array(mdata["rna"].obsm["nhoods"].sum(0)).ravel()
plt.hist(nhood_size, bins=100)
plt.xlabel("# cells in nhood")
plt.ylabel("# nhoods");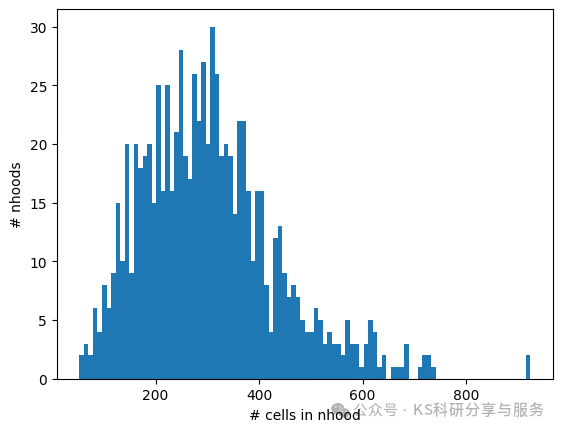
Count cells in neighbourhoods:Milo利用同一实验条件下不同重复样本间的细胞数量变化,来检验(不同组间)差异丰度。因此,需要统计每个样本(样本重复)中的细胞在各个区域的数量。
mdata = milo.count_nhoods(mdata, sample_col="orig.ident")#sample_col就是具有唯一标识的样本id
可视化:可视化的形式与miloR是一致的。
milo.build_nhood_graph(mdata)
plot_nhood_graph(mdata = mdata,
alpha=0.1, ## 显著性阈值,默认0.1
min_size=1, ## 节点size最小值,默认10
min_logFC = 0,#最小logFC值,若该值为 0,则显示所有显著的邻域区域。
plot_edges = True, #是否plot领域图edges,默认False不展示
title = 'Disease_vs_HC DA LogFC' ,#默认是'DA log-Fold Change',可自行修改传入字符串)
color_map = 'viridis')#颜色,python中连续密度颜色谱推荐:viridis,plasma,magma,inf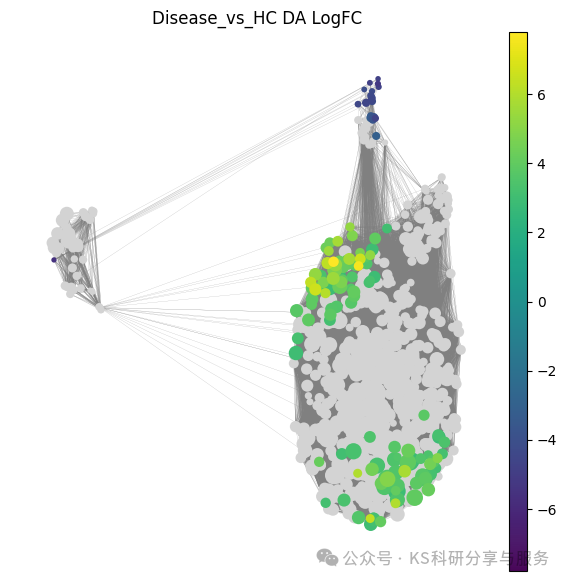
milo.annotate_nhoods(mdata, anno_col="celltype") #anno_col为我们细胞注释标签
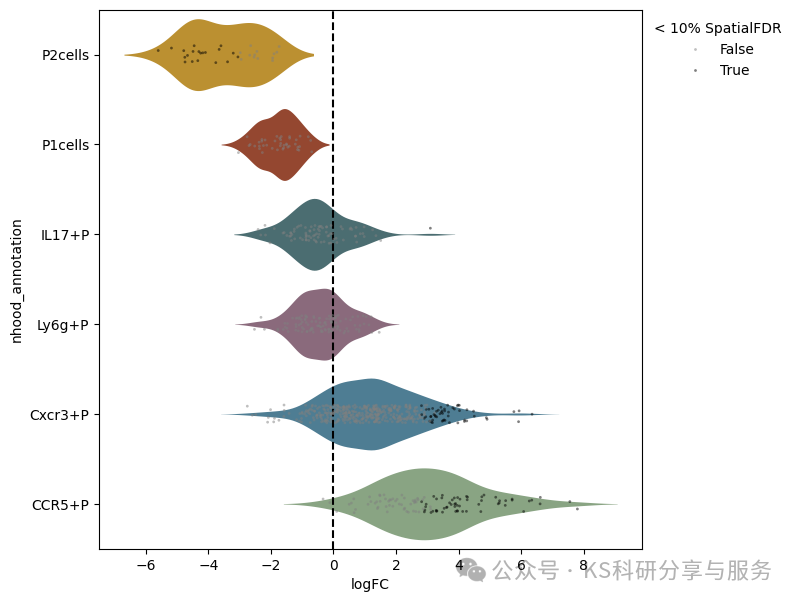
milo.plot_da_beeswarm(mdata,
alpha=0.1,#显著性阈值,默认0.1
palette = ["#d2981a", "#a53e1f", "#457277", "#8f657d", "#42819F", "#86AA7D"],#颜色设置
)
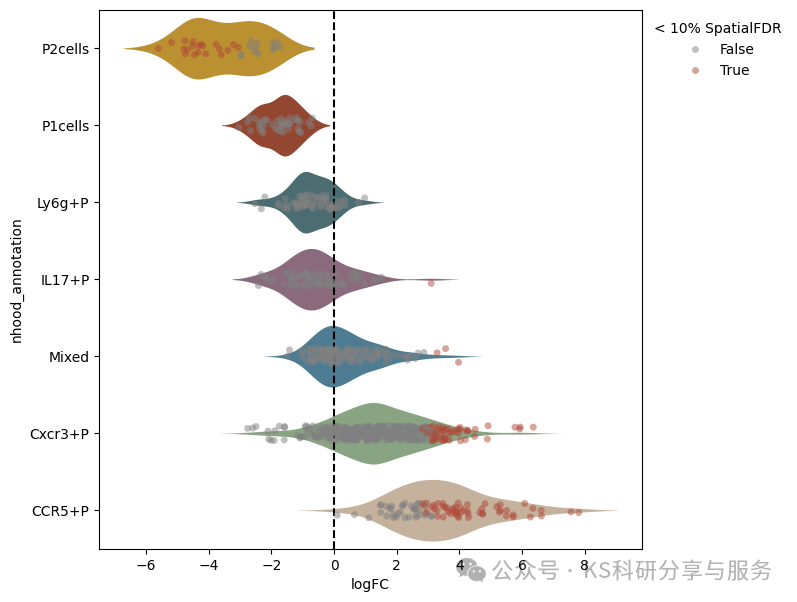
看看每个领域celltype的分布情况,对于大多数区域而言,几乎所有的细胞都具有相同的细胞类型标签。对于那些只有不到 60% 的细胞具有最高标签的区域,我们可以将其重新命名为“混合型”。这里等于是让结果更精准,前面直接可视化的是每个领域最丰富的细胞类型作为领域标签,这里分的更细。
plt.hist(mdata["milo"].var["nhood_annotation_frac"], bins=30)
plt.xlabel("celltype fraction")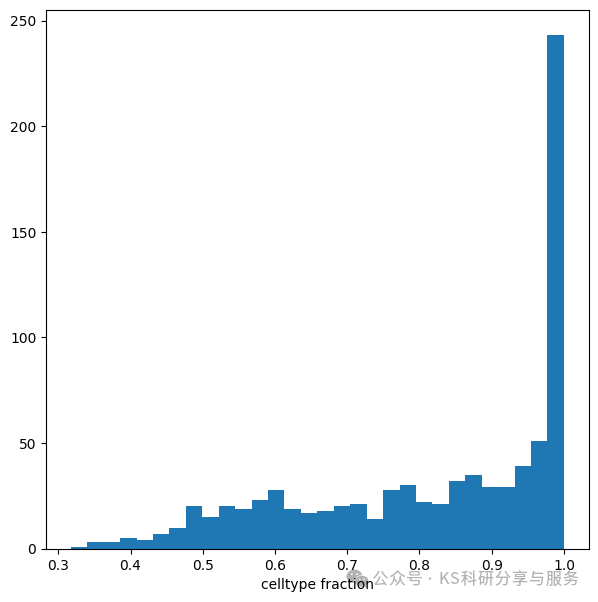
mdata["milo"].var["nhood_annotation"] = mdata["milo"].var["nhood_annotation"].cat.add_categories("Mixed")
mdata["milo"].var.loc[mdata["milo"].var["nhood_annotation_frac"] < 0.6, "nhood_annotation"] = "Mixed"
plot_da_beeswarm(mdata = mdata,#milo data
palette=["#d2981a", "#a53e1f", "#457277", "#8f657d", "#42819F", "#86AA7D","#CBB396"],#颜色设置,函数中celltype标签默认按照logFC从小到大排列
alpha=0.1,#显著性阈值
point_palette = ['grey','#B04838'],#grey是丰度无显著变化的点,'#B04838'是显著变化的,颜色可自行设定
point_size=5)#点的大小