论文解析-scMAGIC
参考
Zhang, Yu & Zhang, Feng & Wang, Zekun & Wu, Siyi & Tian, Weidong. (2022). scMAGIC: accurately annotating single cells using two rounds of reference-based classification. Nucleic acids research. 50. 10.1093/nar/gkab1275.
亮点
- 基于参考数据集的二次分类算法
- 13个对比方法,86个数据集
- 借助已发表的大规模大细胞图谱,可对无参考的单细胞数据进行分类
方法
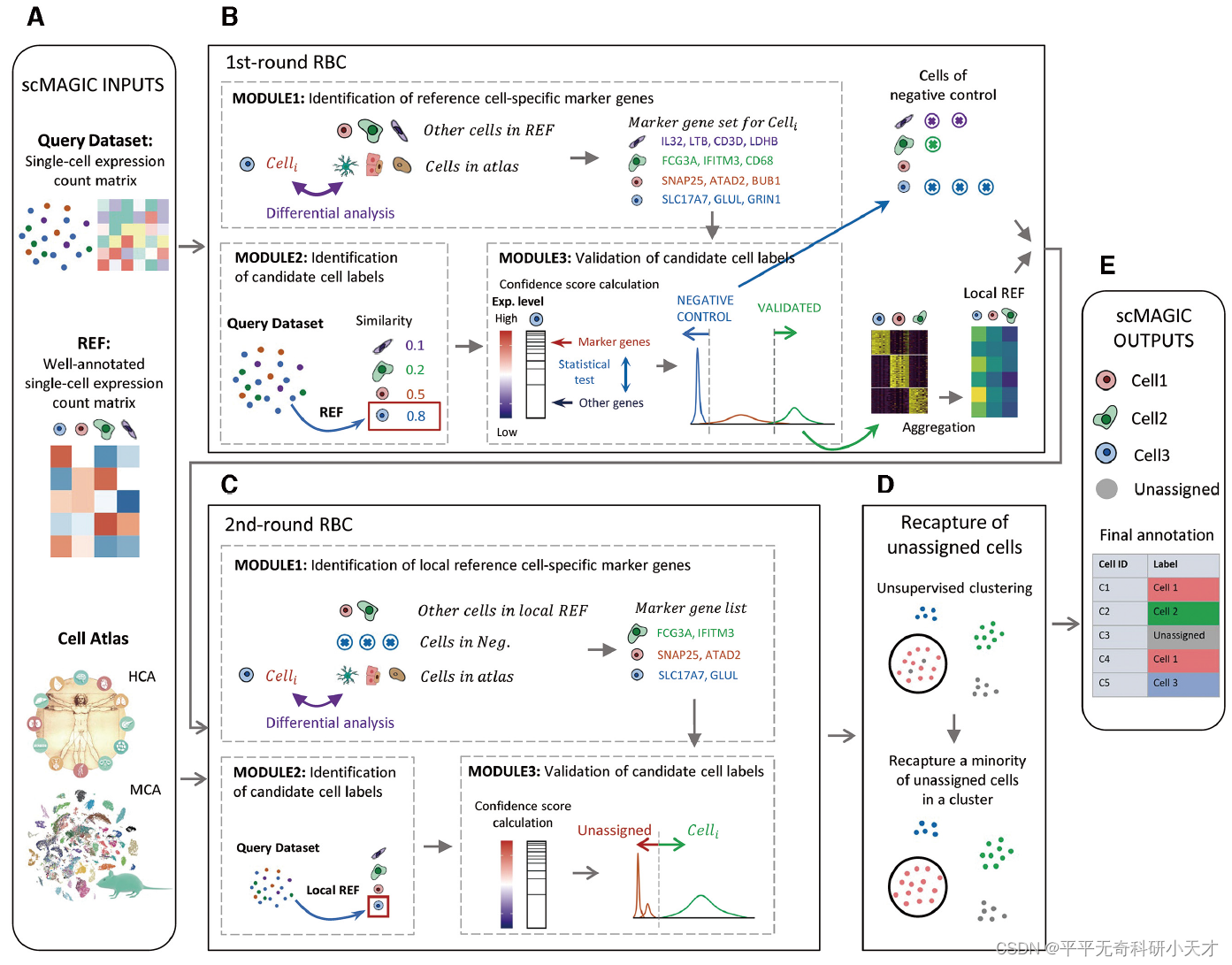
A. 输入
Query的scRNA-seq,Reference的scRNA-seq及标签
同时,该方法内置2个单细胞图谱Atlas(scRNA-seq与对应标签),人类的HCA与MCA
scRNA-seq中行为基因,列为细胞
B. 第一轮RBC
- 识别Reference细胞类型特异性的marker gene
(1)将Reference中同类型细胞的scRNA-seq整合起来
(2)对每种细胞类型,选取前20%高表达的基因A
(3)用limma算法从A中选择相对于其他细胞类型,显著高表达的基因(P value > 0.05)B
(4)用RUVSeq算法消除Reference和Atlas的batch effect
(5)用limma算法从B中选择相对于其他细胞类型,显著高表达的基因(P value > 0.001)C
C即为细胞类型特异性的marker gene,整合了Reference与Atlas的信息。 - 识别候选细胞类型
(1)基因筛选:用Seurat包中‘Find-VariableFeatures’函数选取Reference中高表达的2000个基因
(2)用pcaPP这个R包计算Query中每个细胞与Reference中每个细胞类型的相似度,以Kendall’s tau coefficient作为相似度衡量指标,得到每个细胞的候选细胞类型 - 验证候选类型
(1)将Query根据候选细胞类型分类
(2)对于每一类,通过 Wilcoxon秩和检验(推断两个总体分布的位置是否有差别)检测该类别marker gene是否比其他基因表达显著,通过负对数转换将P value转化为置信度
(3)通过R包mclust将置信度的密度分布分解为多个高斯分布
(4)属于置信度最高的高斯分布的标签,认为是被验证的标签;置信度低于阈值的细胞认为是对应标签的负样本(Negative control)
(5)Negative control将用于第二轮RPC;只利用被验证标签的细胞scRNA-seq构建LocalReference,将作为第二轮RPC的Reference
C. 第二轮RBC
- 识别LocalReference细胞类型特异性的marker gene
(1)将LocalReference中同类型细胞的scRNA-seq整合起来
(2)利用Seurat包中‘Findmarkers’函数,从每一类细胞中识别相对于Negative control,显著高表达的基因A
重复第一轮RBC的(1)-(5),得到的基因即为marker gene - 识别候选细胞类型
(1)以R中‘dmultinom’作为相似度衡量指标,计算Query中每个细胞与LocalReference中每个细胞类型的相似度,得到每个细胞的候选细胞类型 - 验证候选类型
(1)重复第一轮RBC的(1)-(3)
(2)计算每个高斯分布的均值:如果均值低于阈值(5),则该分部内的细胞预测标签为‘unassigned’;否则,将细胞的候选类型作为预测结果
D. 对无标签细胞进行无监督聚类
(1)利用Seurat包中‘FindClusters’函数对Query中所有细胞聚类
(2)如果一个簇中大多数细胞都被预测为同一类别,且其中‘unassigned’细胞占比低于20%,则认为这些无标签细胞与该簇的其他细胞为同一类别。
数据集
| Dataset | Tissue | Reference | Link |
|---|---|---|---|
| Mouse brain dataset | primary visual cortex(PVC) | Tasic et al. | GSE71585 |
| primary somatosensory cortex (S1) | Zeisel et al. | GSE60361 | |
| hippocampal CA1 region | GSE115746 | ||
| neocortex | Tasic et al. | GSE93374 | |
| hypothalamic arcuate-median eminence complex (HArcME) | Campbell et al. | GSE95315 | |
| dentate gyrus of the hippocampus (DGH) | Hochgerner et al. | GSE109447 | |
| ventricular-subventricular zone (V-SVZ) | Mizark et al. | ||
| Mouse pancreas dataset | Baron et al. | GSE84133 | |
| Mouse duodenum dataset | Haber et al. | GSE92332 | |
| Human pancreas datasets | Baron et al., Muraro et al., Xin et al., Segerstolpe et al. | GSE84133, GSE85241, GSE81608, E-MTAB-5061 | |
| Human PBMC datasets | Ding et al., Butler et al., Zheng et al. | GSE132044, Seurat Data, https://support.10xgenomics.com/single-cellgene-expression/datasets | |
| CellBench 10X dataset | mixture of five human lung cancer cell lines | Tian et al. | GSM3618014 |
| CellBench 10X dataset | mixture of five human lung cancer cell lines | Tian et al. | GSM3618022, GSM3618023, GSM3618024 |
| Tabula Muris dataset | The Tabula Muris Consortium | GSE109774 | |
| MCA Atlas | GSE108097 | ||
| HCL Atlas | GSE134355 |
评价指标
- Accuracy
类别预测正确的细胞数占细胞总数的比例 - Balanced Accuracy
分别计算每个类别的Accuracy,再平均 - Cluster accuracy
(1)对于Query中每个细胞,用Seurat包中‘FindClusters’函数进行无监督聚类
(2)用本文方法或对比方法,为Query中每个细胞预测Label
(3)对于无监督聚类的每个簇,以其中数量最多的Label作为该簇的类别
(4)计算类别预测正确的聚类数占聚类总数的比例,即为Cluster accuracy
对比算法
| Category | Method | Reference |
|---|---|---|
| Statistical metric based methods | SciBet | Li,C., Liu,B., Kang,B., Liu,Z., Liu,Y., Chen,C., Ren,X. and Zhang,Z.(2020) SciBet as a portable and fast single cell type identifier. Nat.Commun., 11, 1818. |
| SingleR | Aran,D., Looney,A.P., Liu,L., Wu,E., Fong,V., Hsu,A., Chak,S.,Naikawadi,R.P.,Wolters,P.J., Abate,A.R. et al. (2019)Reference-based analysis of lung single-cell sequencing reveals a transitional profibrotic macrophage. Nat. Immunol., 20, 163–172. | |
| scmap | Kiselev,V.Y., Yiu,A. and Hemberg,M. (2018) scmap: projection ofsingle-cell RNA-seq data across data sets. Nat. Methods, 15, 359–362. | |
| Tree-based methods | CHETAH | de Kanter,J.K., Lijnzaad,P., Candelli,T., Margaritis,T. and Holstege,F.C.P. (2019) CHETAH: a selective, hierarchical cell type identification method for single-cell RNA sequencing. Nucleic Acids Res., 47, e95. |
| scClassify | Lin,Y., Cao,Y., Kim,H.J., Salim,A., Speed,T.P., Lin,D.M., Yang,P. and Yang,J.Y.H. (2020) scClassify: sample size estimation and multiscale classification of cells using single and multiple reference. Mol. Syst. Biol., 16, e9389. | |
| Machine-learning-based methods | SingleCellNet | Tan,Y. and Cahan,P. (2019) SingleCellNet: a computational tool to classify single cell RNA-Seq data across platforms and across species. Cell Syst., 9, 207–213. |
| scPred | Alquicira-Hernandez,J., Sathe,A., Ji,H.P., Nguyen,Q. and Powell,J.E. (2019) scPred: accurate supervised method for cell-type classification from single-cell RNA-seq data. Genome Biol., 20, 264. | |
| scID | Boufea,K., Seth,S. and Batada,N.N. (2020) scID uses discriminant analysis to identify transcriptionally equivalent cell types across single-cell RNA-Seq data with batch effect. Iscience, 23, 100914. | |
| CaSTLe | Lieberman,Y., Rokach,L. and Shay,T. (2018) Correction: castle - Classification of single cells by transfer learning: harnessing the power of publicly available single cell RNA sequencing experiments to annotate new experiments. PLoS One, 13, e0208349 | |
| SVM | Pedregosa,F., Varoquaux,G., Gramfort,A., Michel,V., Thirion,B., Grisel,O., Blondel,M., Prettenhofer,P., Weiss,R., Dubourg,V. et al. (2011) Scikit-learn: machine learning in python. J. Mach. Learn Res., 12, 2825–2830. | |
| semi-supervised learning methods | Seurat v4 | Hao,Y., Hao,S., Andersen-Nissen,E., Mauck,W.M. 3rd, Zheng,S., Butler,A., Lee,M.J., Wilk,A.J., Darby,C., Zager,M. et al. (2021) Integrated analysis of multimodal single-cell data. Cell, 184, 3573–3587. |
| scSemiCluster | Chen,L., He,Q., Zhai,Y. and Deng,M. (2021) Single-cell RNA-seq data semi-supervised clustering and annotation via structural regularized domain adaptation. Bioinformatics, 37, 775–784. | |
| CALLR | Wei,Z. and Zhang,S. (2021) CALLR: a semi-supervised cell-type annotation method for single-cell RNA sequencing data. Bioinformatics, 37, i51–i58. |
实验评估
3种经典分类场景
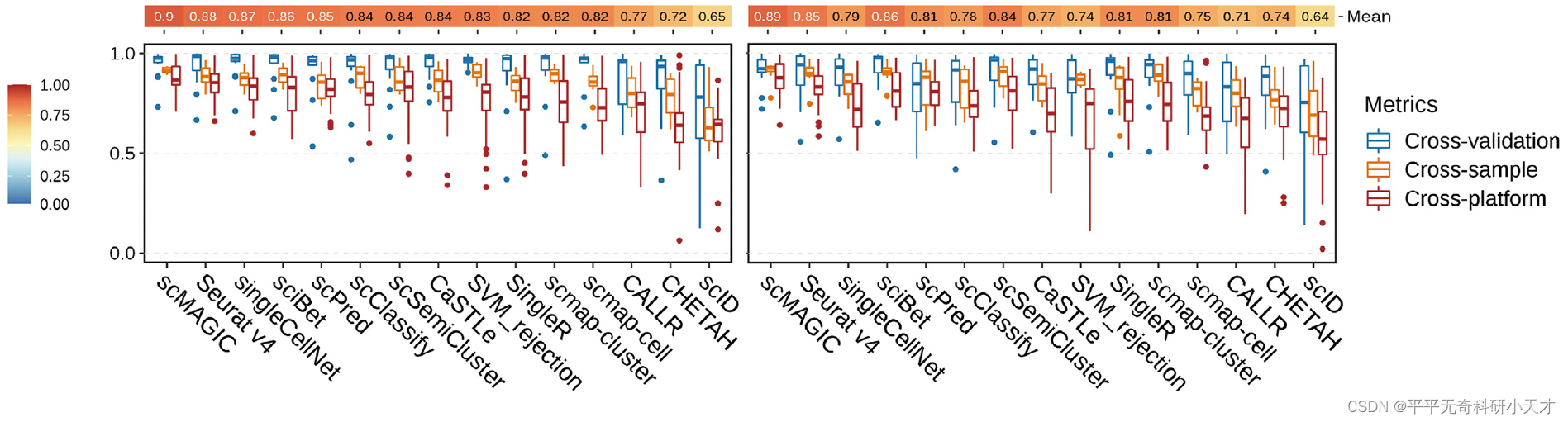
1. Reference包含Query中所有细胞类型
此处模拟Reference和Query之间不同程度的BatchEffect,分别考虑同一数据集的五折交叉验证(无BatchEffect),相同平台相同组织的不同样本数据集(小BatchEffect),不同平台数据集(大BatchEffect)。左列为Accracy值,右列为BalancedAccuracy值。scMAGIC结果达到0.9左右。
2. Reference包含Query中部分细胞类型
对于Query中未出现在Reference的细胞类型,他们的正确Label为‘unassigned’。同时,为了模拟不同程度的BatchEffect,分别考虑深度测序场景(Reference的scRNA-seq中平均每个细胞包含超过2500个基因,Smart-seq2等)代表小BatchEffect,低度测序场景(Reference的scRNA-seq中平均每个细胞包含低于650个基因,Seq-Well等)代表大BatchEffect。左列为Accracy值,右列为BalancedAccuracy值。scMAGIC结果达到0.9左右。
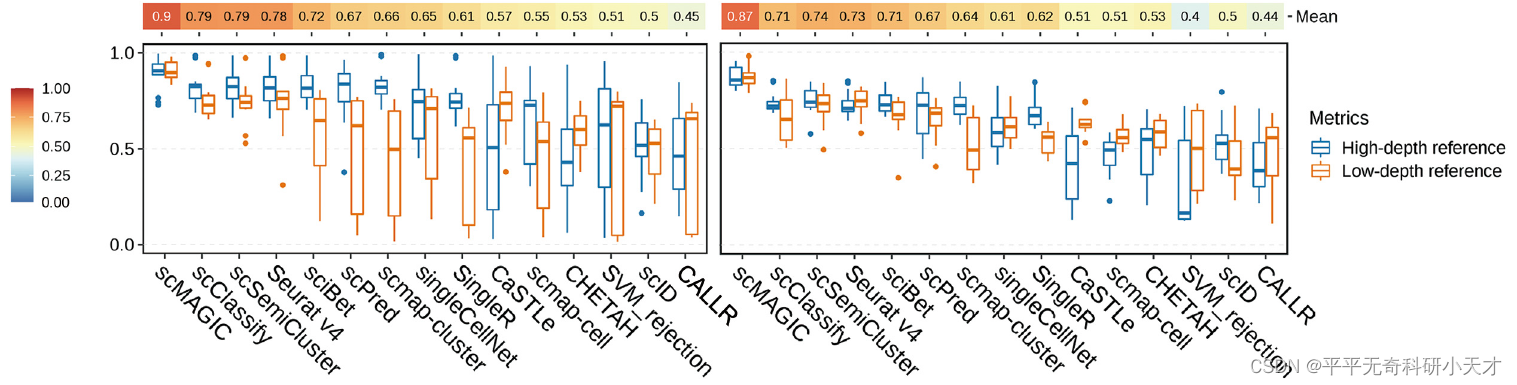
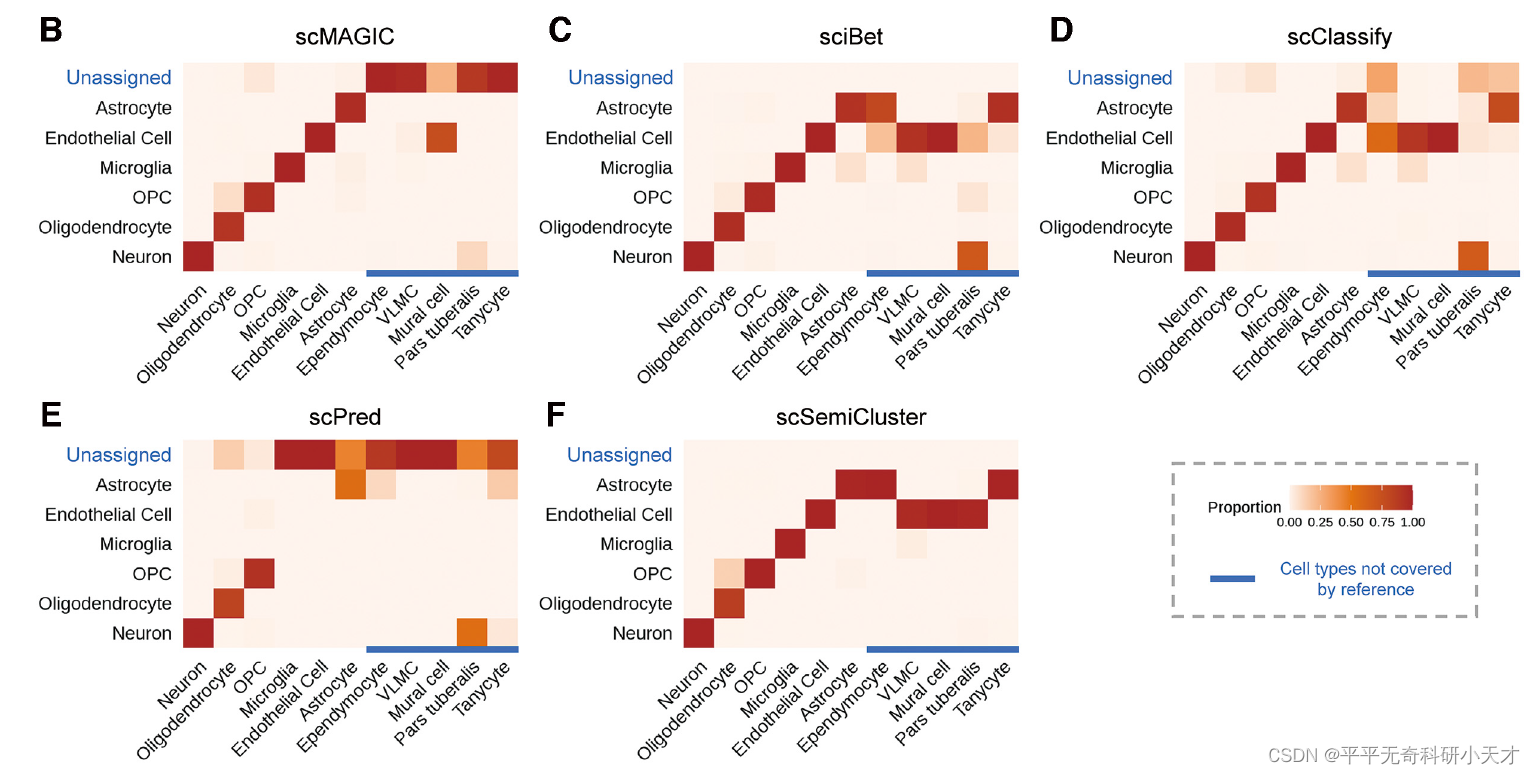
以小鼠大脑数据集作为CaseStudy。行为Query真实类别,其中后5种未包含在Reference中,因此其正确预测Label应为‘Unassigned’。对比结果看出,scMAGIC做出的预测最接近真实Label,scPred为第二名。
3. 跨物种的细胞类型识别
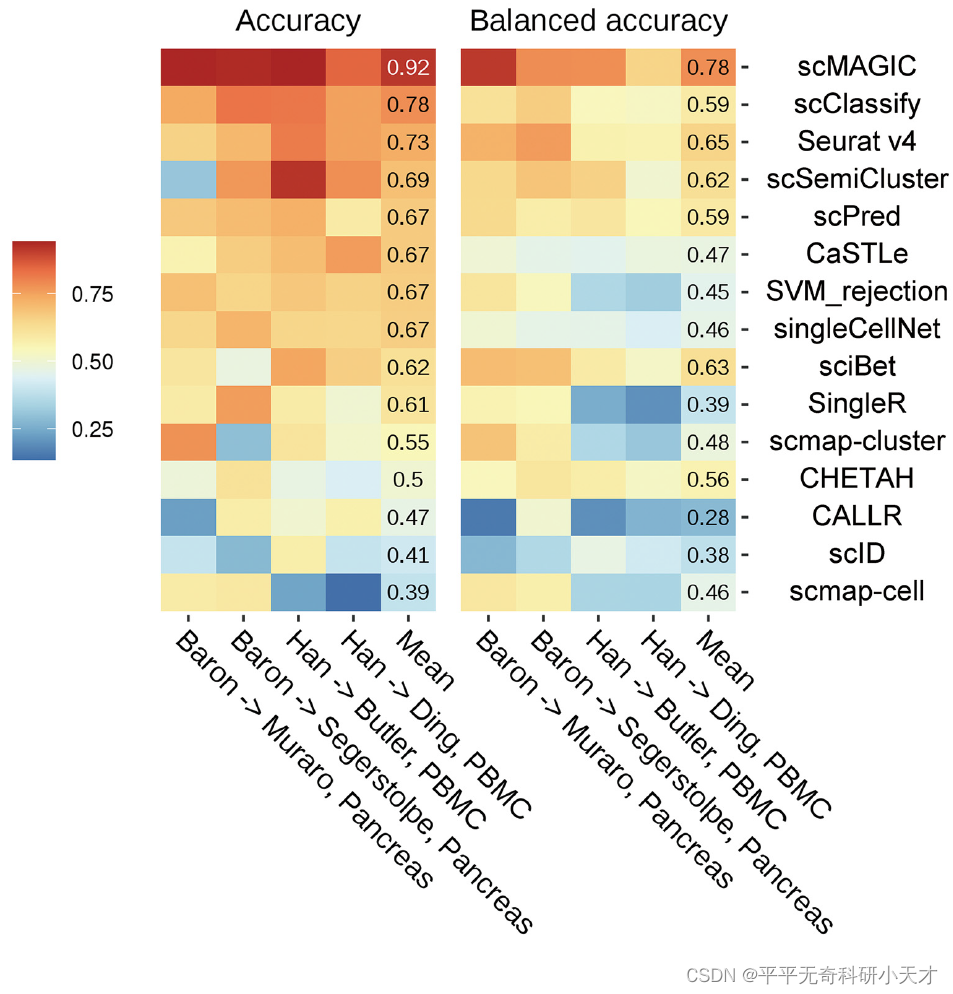
对于同一组织,以小鼠数据集作为Reference,人类数据集作为Query。通过基因同源性进行基因对齐,因此存在很大BatchEffect。结果表明,scMAGIC的Accuracy达到0.92,Balanced accuracy达到0.78.(跨物种预测比前2种同物种场景下的Accuracy高???)
方法在所有场景的综合评估
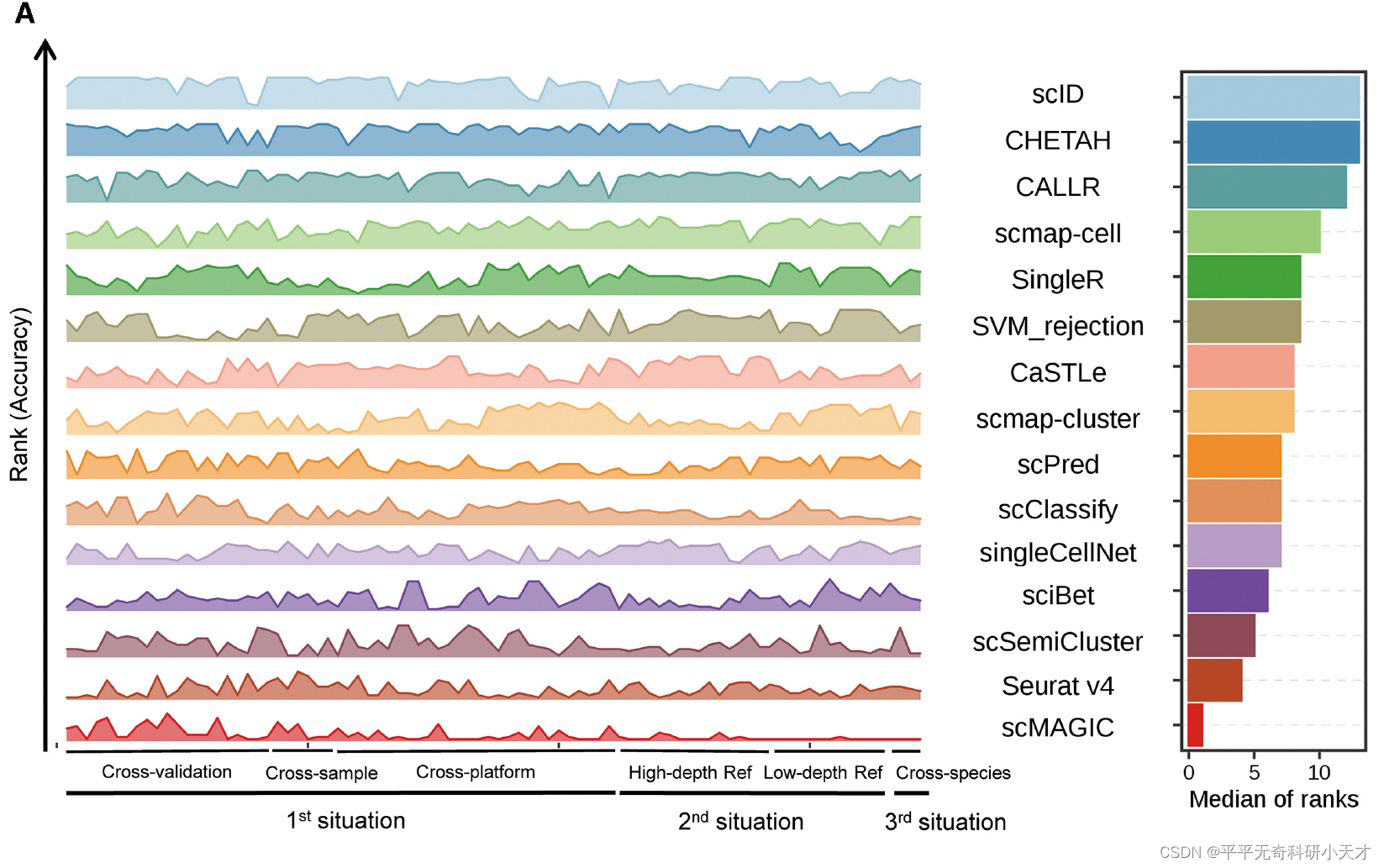
横坐标为不同实验场景,纵坐标为每种方法在对应实验中的排名。左图看出在交叉验证中,大多数方法都很好,scMAGIC并不突出;但在低测序深度与跨物种预测的场景下,大多数方法效果均不好,scMAGIC优势明显。右图看出,综合所有场景,scMAGIC性能稳定,综合准确性排名第一。
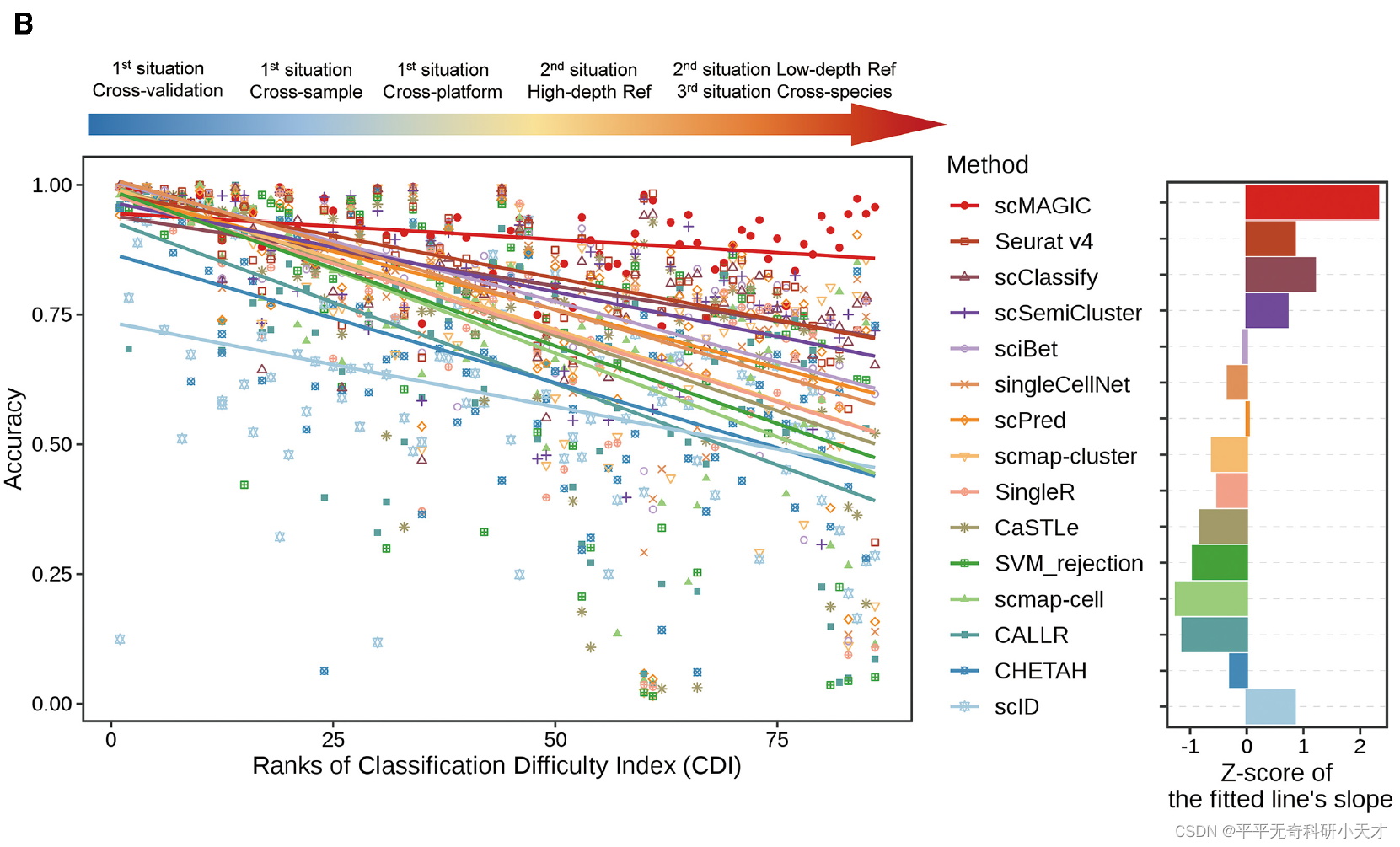
此处利用指标CDI(考虑Reference和Query之间的BatchEffect)衡量预测任务的难度,横坐标为所有实验场景的难度系数,纵坐标为各方法在各实验场景下的Accuracy,直线为每种方法所有点的线性回归。左图表明所有方法准确率随着任务难度增大而降低,其中scMAGIC对于低难度分类任务没有优势,但对于高难度分类任务优势明显。
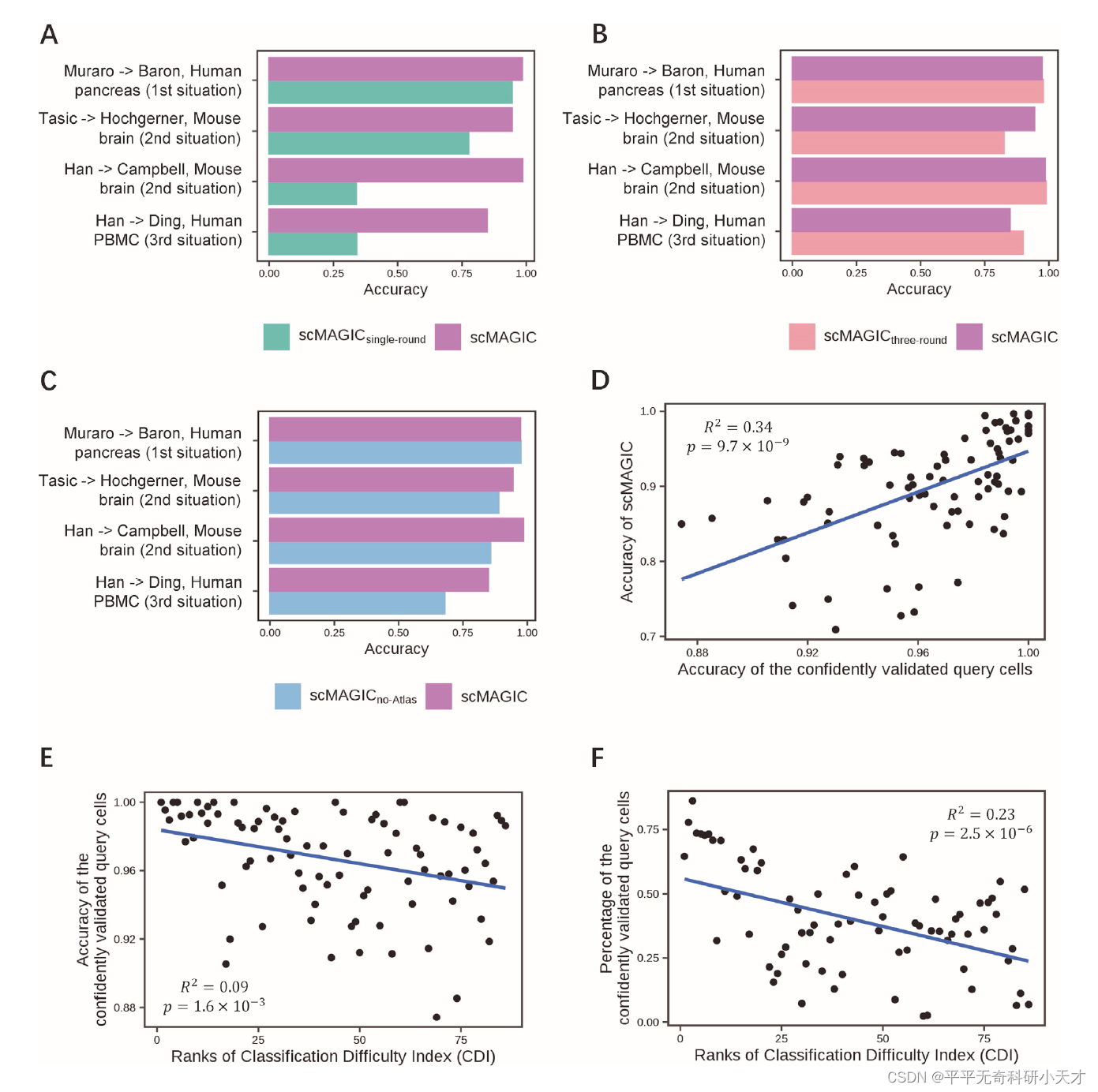
评估scMAGIC中各模块的重要性
A:分别在不同场景数据集中,测试第二轮RBC的重要性,即LocalReference的作用。结果表明,在BatchEffect较小情况下,LocalReference作用不明显;在BatchEffect较小情况下,LocalReference替换Reference后能明显提高Accuracy。
B:分别在不同场景数据集中,测试第三轮RBC的必要性。结果表明,第三轮RBC对预测精度提升不明显。这是由于第二轮RBC已经引入高质量的LocalReference,第三轮没有引入更多有效信息,所以不需要进行第三轮RBC。
C:分别在不同场景数据集中,测试Atlas的重要性。结果表明,在BatchEffect较大情况下,Atlas能明显提高Accuracy。因为Atlas引入很多额外数据及标签,相当于增加训练集,当然明显提升Accuray,尤其是跨物种,大数据带来高精度。
D:评估LocalReference的可信度对预测结果的影响。横坐标为第一轮RBC后被验证细胞的Accuracy,纵坐标为最终预测Accuracy。结果表明,LocalReference的Accuracy与最终预测结果的Accuracy正相关,可信度高的训练集导致准确的预测结果。
E:评估分类难度对LocalReference可信度的影响。结果表明,分类难度与LocalReference的Accuracy负相关,合理。
F:评估分类难度对LocalReference包含细胞数量的影响。结果表明,分类难度与LocalReference大小负相关,即困难的分类任务具有较高置信度的预测结果数量较少,因为每个样本都更加难以分类。
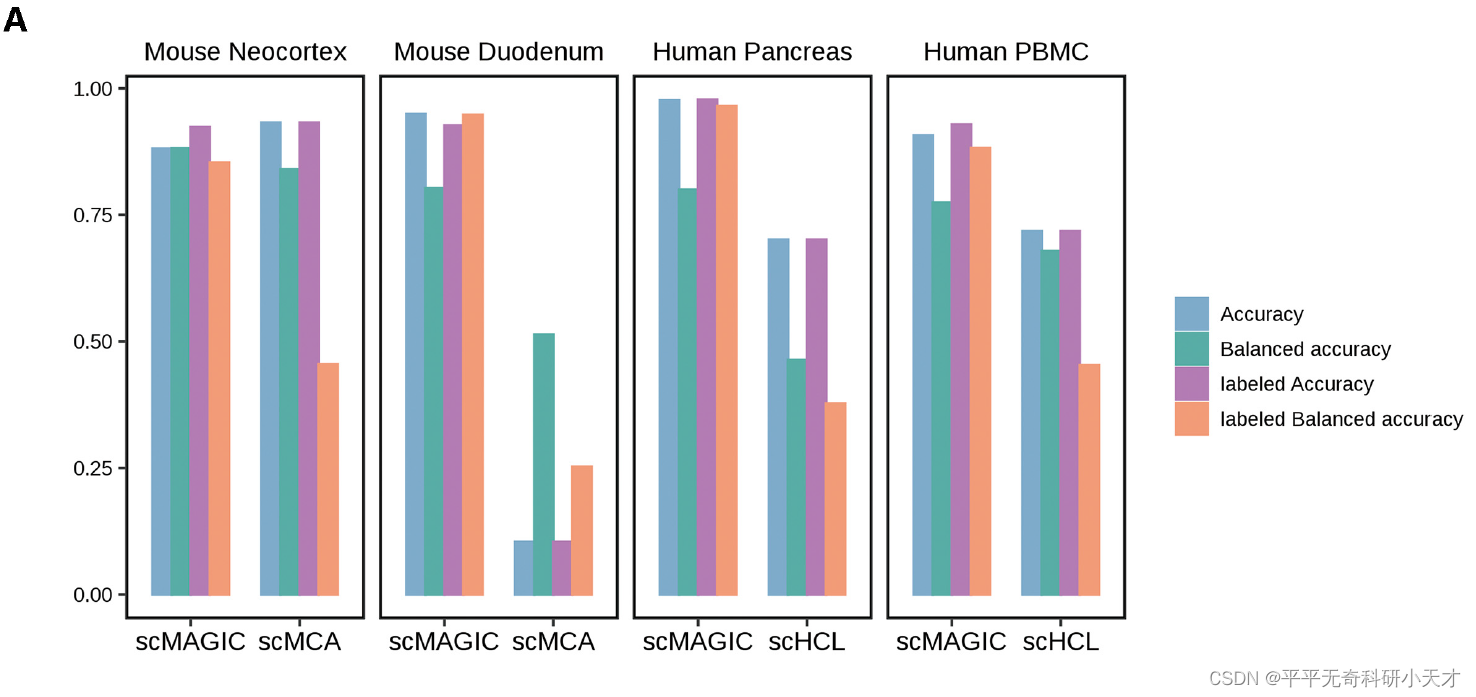
scMAGIC可作为无参考数据集的细胞注释工具
scMAGIC根据内置的MCA和HCL两个Atlas,即可在researcher不提供Reference的情况下对Query做出高准确度的细胞注释。
scMCA与scHCL是MCA和HCL文献中提出的细胞注释方法。结果表明scMCA对一些小鼠数据集注释的Accuracy很低,scHCL对一些人类数据集注释的Accuracy不高。scMAGIC在researcher不提供Reference的情况下,在该4个数据集的平均预测Accuracy可达到0.92-0.98,因此可作为十分可靠的细胞注释工具。